▼もくじ [非表示]
がん概論
イントロダクション
20世紀の間、科学者はがん増殖を促進する細胞および分子のメカニズムを学びました。過去50年にみられる生物医学的研究の加速で、予測可能な方法で細胞機能を変更することが可能となり、疾患の検出および革新的な研究方法が科学界および医学界に提供され、がんなどの重篤な病状を治療するための新しい方法を見つけることが可能となりました。
本記事では、はじめに、がん増殖シグナル伝達の研究を可能としてきたいくつかの重要なマイルストーンについて言及します。次に、がん研究の進歩において可能となった、がん標的治療の実用化の具体的な方法を図解入りでご紹介いたします。最後に、ウェスタンブロット、免疫蛍光分析、フローサイトメトリー、ELISA、がん生物学および他の領域の生物医学的研究に常用されている幅広い実験技術の詳細なプロトコールをご紹介いたします。
一般統計
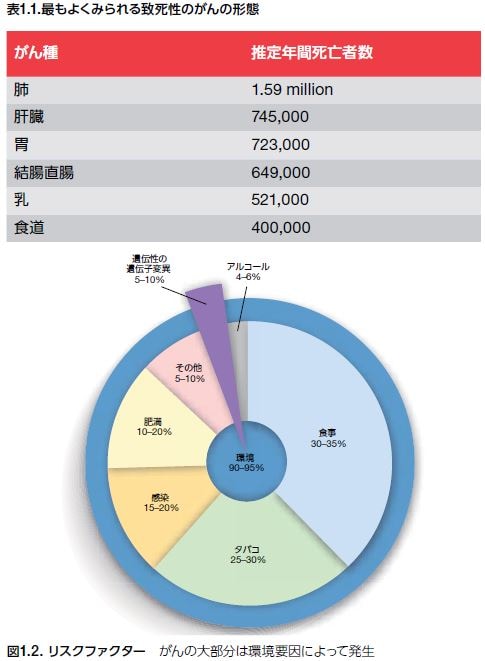
「がん」は、重要な細胞機能を制御不能とする特定の遺伝子変異を特徴とする疾患群を表す一般的用語です(図1.1)。がんは、体内のあらゆる器官に発生する可能性があり、固形腫瘤を形成するか、あるいは血液系の細胞を侵す腫瘍を形成するとされています。「悪性腫瘍」および「新生物」は、「がん」を示す別の用語です。全てのがんは、起源の組織に関わらず、細胞増殖の加速、解剖異常細胞の存在、体内の他の組織に転移または浸潤するという共通の特徴を示します。転移性疾患はがん末期に生じ、がん罹患者の主要な死亡原因となっています。世界保健機関によれば、がんは世界的に主要な死亡原因で、2012年のがん関連の死亡者数は820万人と発表されています。いくつかの一般的ながん種に関連する世界における推定年間死亡者数を表1に示します*1。
医学界では、全てのがんの5%~10%が遺伝性であると認識されています(図1.2)。残りの90%~95%のがんは、毒素(タバコなど)、UV照射および他の環境要因、不健康な食事や過剰なアルコール摂取などの生活要因を始めとするさまざまな原因によって生じる遺伝子変異によって発生し、多くのさらなる要因によって、がんが発現するリスクが高まると考えられています*1,2。
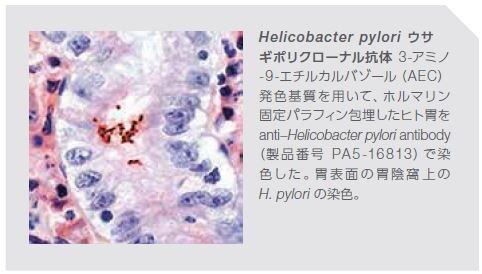
他のがん関連リスクファクターとして、特定のウイルスおよび細菌による感染があります。例えば、子宮頸がんおよび胃がんが発生する可能性は、それぞれヒトパピローマウイルスおよびHelicobacterpylori(ピロリ菌)による感染によって上昇します。Helicobacterpylori は、ヒトにおける消化性潰瘍および慢性胃炎の原因としてよく知られています。この細菌は十二指腸潰瘍にも関連し、腺がんおよび胃における粘膜関連リンパ組織の低悪性度リンパ腫の発生にも関与すると考えられています*3,4。
当記事は、がん増殖シグナル伝達経路の探索ガイドブックからの抜粋です。
ガイドブックは下記から無料でダウンロード頂けます。
がん研究マイルストーン
注目すべき発見
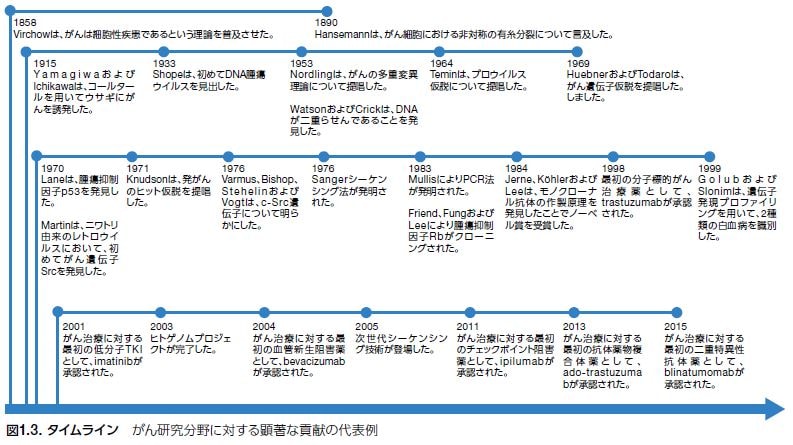
1940年代から1950年代に研究者は、がんの発症には遺伝的背景が関与している可能性があると仮定し始めました*6 。そのような考えは、現代の分子がん研究を立ち上げたパラダイムシフトを代表するものです。がん研究の発展を支援し、腫瘍増殖を阻害する可能性を持つ分子標的治療の開発を促進してきた発見の代表例を図1.3に示します*5–13。
発がんの2ヒット説
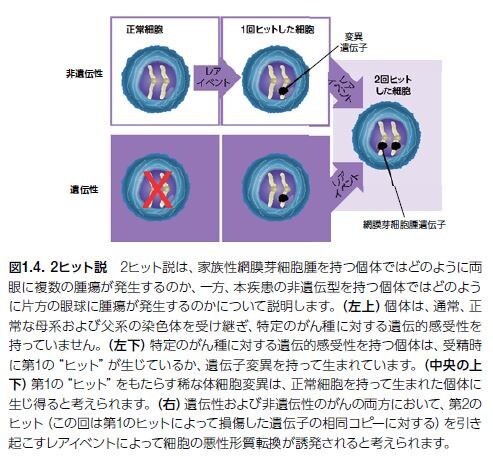
1950年代の間は疫学データおよび数学的モデルに依存していましたが、Nordlingらは、がんは連続的な変異(すなわちヒット)によって発生すると提唱しました。これにより、高齢化に伴いさまざまなヒトがんの発生頻度が増加することの説明が可能です。Knudsonは、がんの発症に関与するヒットの最小数について理解することを求めて、通常幼児期に発症する網膜の腫瘍である網膜芽細胞腫(RB)の根底にある遺伝的メカニズムについて調査する研究を開始しました(図1.4)*14,15。
Knudsonの統計解析から、疾患には2つのヒット、すなわち変異が関与する2種類の形態が存在することが予測されました。遺伝性網膜芽細胞腫のケースでは、最初のヒットは受精時に存在する遺伝子変異に関係していたのに対し、それに続く第2のヒットは同じ遺伝子の他方のコピーに生じる体細胞変異でした。家族性網膜芽細胞腫を受け継いだ子供が第2のヒットを発生する可能性は、変異を持たない子供の100,000倍以上です。逆に言えば、このモデルから、網膜芽細胞腫を生じる遺伝子を有する疑いのない個体において疾患が生じるには、2つの体細胞変異(類似した変異率で発生する)が必要であることが予測されました*16。
1971年に、間接的に腫瘍抑制遺伝子の概念に繋がる研究が公開され*17、 発がんの2ヒット説(Knudson仮説)が提唱されました。これは、網膜芽細胞腫を含む数種の主要ながんにおいて見られる変異であるRB1遺伝子が発見される15年前で、初めてヒト染色体の配列決定が行われた約28年前のことでした*17,18。
これまでに複数のヒト腫瘍抑制遺伝子(いくつかは家族性がん症候群およびがんの非遺伝型)がクローニングされています。同定された新しい候補遺伝子が既知腫瘍抑制因子のリストに追加され、調査および検証されていくものと考えられます*19 。
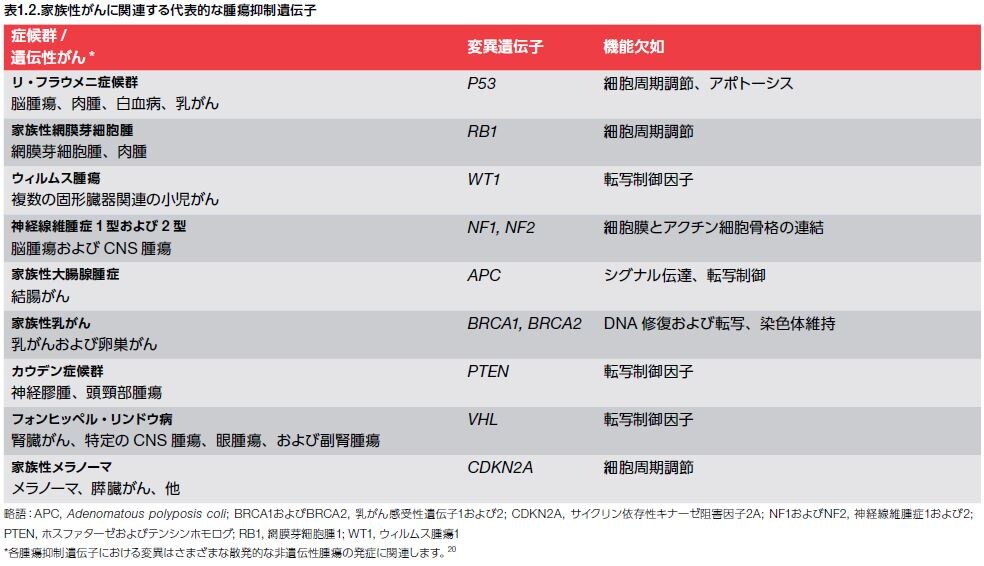
RB1変異に関連するがんの高い発生率は、この腫瘍抑制遺伝子が重要であることを浮き彫りにしています。RB1の機能喪失は、小児における網膜芽細胞腫に関連します。RB1遺伝子によってコードされるタンパク質であるpRbは、細胞周期の制御因子であり、RB経路の調節不全はヒトがんの大部分の形態において観察されます*21。 家族性がんに一般的に関連する腫瘍抑制因子の例を表1.2に示します*17,20。
がんゲノム研究
がんのホールマーク(特徴)
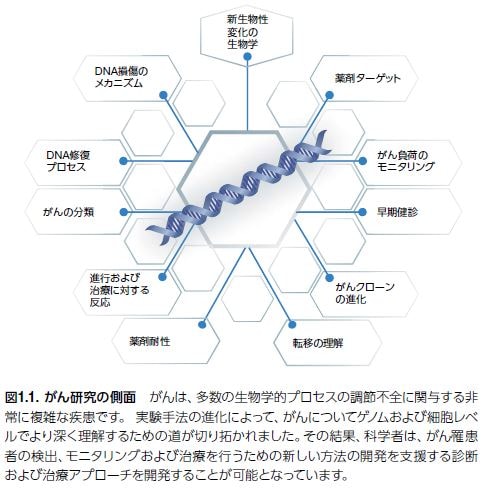
がん研究者は、DNA増幅およびシーケンシング技術における大きな進展とともに、腫瘍形成過程に生じる細胞および分子の変化ならびに遺伝的不安定性に関する詳細な情報を提供してきました。がんゲノム解析は、腫瘍形成開始、がん進行、転移および薬剤耐性に関連する特定の変異の同定および機能的分類を可能にしてきました。22 大規模な研究によって、腫瘍形成過程(増殖、エピジェネティックな修飾、成長および代謝、アポトーシス、ならびに他のプロセス)に異常性が発揮されるさまざまな細胞プロセスが同定されました。同様に、慢性炎症、病的血管新生、および免疫系回避は、集合的にがん進行および転移を促進します*23。
HanahanおよびWeinbergの提唱するがんのホールマークは、腫瘍細胞が示す特徴的で補完的な能力を表し、新生物疾患の複雑さを概念的に説明するフレームワークを提供します(図1.5)。これらの組織化された原理に焦点を当てることは、新しい研究分野を探索し、がん患者のために新しい治療方針を考案できるように研究者を支援することにつながります*24。
がん:体細胞変異の蓄積
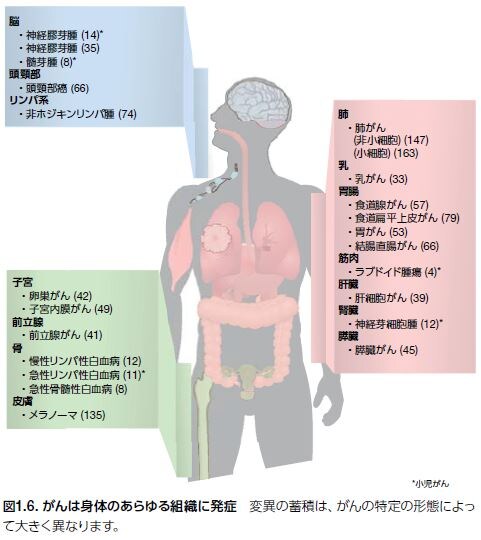
成人および小児腫瘍の多様なアレイのゲノムワイドなシーケンシング研究により、さまざまなヒトがんに関連する体細胞変異の数およびタイプについての情報が得られました(図1.6)*25 。
例えば、身体のさまざまな組織や細胞に生じる体細胞変異の数は、小児ラブドイド腫瘍に見られるわずか4変異から、小細胞肺がんに見られる163変異までに及びます。乳房、結腸、脳、ならびに他の器官および組織に発生する一般的な固形がんでは、タンパク質産物を変化させるわずかな体細胞変異の平均数は33から66です。これらの変異の95%以上は、1塩基置換です。約5%の変異では、欠失、転座、または挿入が観察されます*25。
正常細胞において生じる点変異の割合は、腫瘍細胞において生じる点変異の割合に反映されます。しかしながら、がん細胞では染色体変化の割合が上昇します。これらの変異では、染色体の数、欠失、転座、遺伝子増幅、および他の異常において変化が生じます *26。
がんのドライバー変異およびパッセンジャー変異
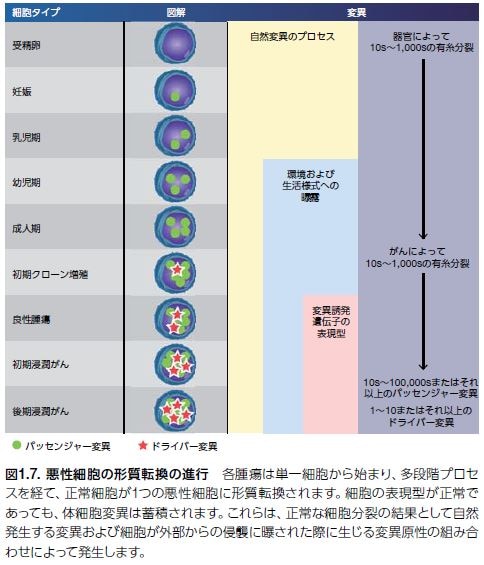
がん細胞で検出される点変異および転座において、遺伝子変異の大部分は腫瘍細胞の適応性(すなわち選択的増殖優位性)に影響しません(図1.7)*26,27 。
定義では、正常細胞よりも腫瘍細胞に対し増殖優位性を与えることがない変異はパッセンジャー変異と考えられます。一方、正常細胞よりも腫瘍細胞に対し増殖優位性を与える遺伝子はドライバー変異と考えられます。ドライバー遺伝子の中には、ドライバー変異が含まれます。しかしながら、ドライバー遺伝子にはパッセンジャー変異も含まれ得ることに留意することは重要です *25,28。
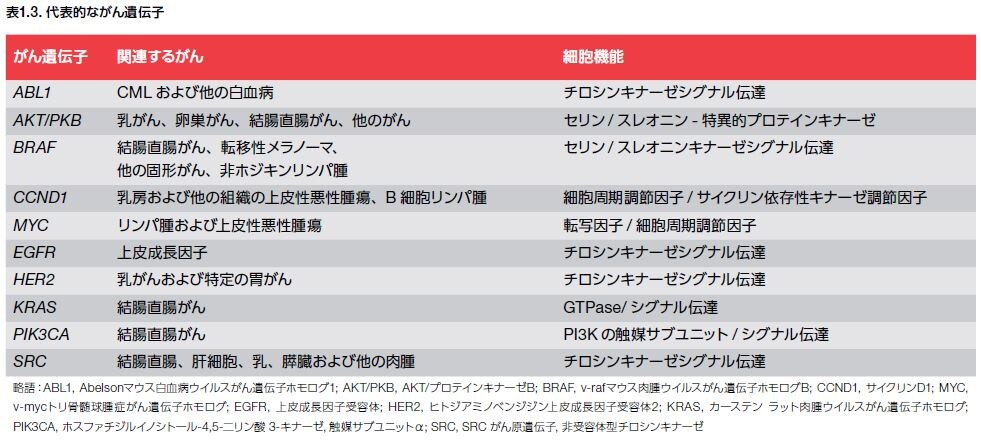
 腫瘍抑制遺伝子の変異と同様に、発がん性変異はドライバー変異として分類されます *29。 がん遺伝子は、正常な細胞プロセスを調節するがん原遺伝子に由来しますが、がん原遺伝子における機能獲得型変異は、腫瘍への形質転換および増殖を誘発する可能性を持つ異常な遺伝子産物を産生します(図1.8)。代表的ながん遺伝子については、表1.3を参照してください *33–35 。
腫瘍抑制遺伝子の変異と同様に、発がん性変異はドライバー変異として分類されます *29。 がん遺伝子は、正常な細胞プロセスを調節するがん原遺伝子に由来しますが、がん原遺伝子における機能獲得型変異は、腫瘍への形質転換および増殖を誘発する可能性を持つ異常な遺伝子産物を産生します(図1.8)。代表的ながん遺伝子については、表1.3を参照してください *33–35 。
ドライバー変異とパッセンジャー変異を同定および識別することを目的とした、大規模なゲノムデータセットを取得し解釈する能力は、がんゲノム解析に従事する研究者の重要な目標です。ドライバー遺伝子および変異について定義および検証する能力は、がんの発生、進行および薬剤耐性に関するメカニズムをさらに解明する助けとなります *30。
がん遺伝子概論
数十年にわたる研究の後、1970年にSRCが発見され、初めてがん遺伝子が同定されました。この遺伝子は、ニワトリにおいてがんを引き起こすことで知られるラウス肉腫ウイルスのv-src遺伝子に非常に類似しています。このがん原遺伝子は、胚発生および細胞成長を調節する役割を果たしていると考えられます。SRCによってコードされるタンパク質は、チロシンプロテインキナーゼで、その活性はC末端Srcキナーゼ(CSK)によるリン酸化によって阻害されます。*32,33 さまざまな動物種およびヒトにおいて、SRC変異は、結腸、肝臓、肺、乳房、および膵臓のさまざまながんの悪性化進展に関与しています *34–36。
当記事は、がん増殖シグナル伝達経路の探索ガイドブックからの抜粋です。
ガイドブックは下記から無料でダウンロード頂けます。
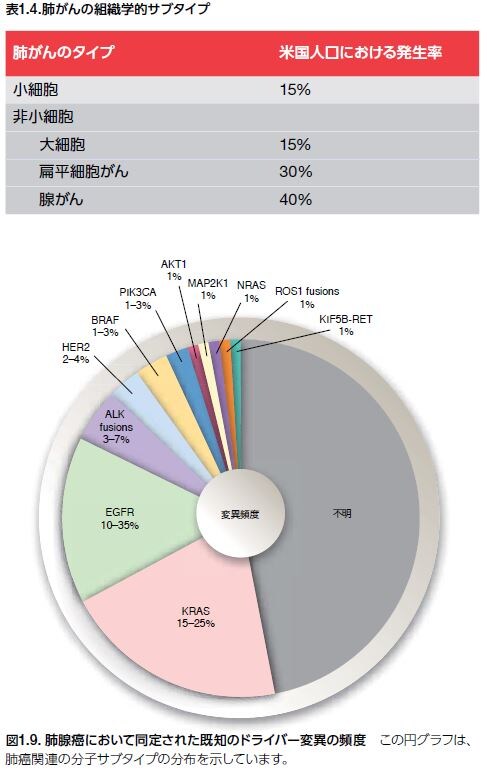
肺癌のサブタイプ
肺がんのドライバー変異
肺がんは世界的にがん関連死の原因の第1位で、喫煙と密接に関係しています。これまでに、これらの治療困難ながんに関係するいくつかの臨床的に関連するドライバー変異が同定されています *37–40 。
肺がんゲノム解析は、組織学的サブタイプデータと分子生物学的サブタイプデータを結びつける手段として、がん生物学者および腫瘍学者に提供されています(表1.4および図1.9)。これらの近年の見識が、診断目的̶および現在増加してきている個々の患者の治療目的に適用されることによって、よりパーソナルな治療アプローチの開拓が続けられています *37–40 。 例えば、2013年には、8つの個別の臨床試験において、上皮成長因子受容体(EGFR)遺伝子変異陽性非小細胞肺がん(NSCLC)患者を対象とした、EGFR阻害剤と従来の化学療法の比較が行われました。各試験の結果から、この患者集団において、EGFR阻害剤は化学療法と比較して、複数のパラメータにおいて一貫して優れていることが示されました。NSCLC患者の標的治療は、奏効率、生活の質(QOL)、および無増悪生存率において統計学的に有意な改善を示しました。しかしながら、これらの臨床試験結果は、EGFRチロシンキナーゼ阻害剤(TKI)を投与した患者における明確な延命効果を示すことはできませんでした。研究者は、有意な治癒率の達成を目指して、NSCLCに対する標的治療と併用する補助療法の開発に引き続き焦点を当てています *40,41。
肺がんにおける標的治療
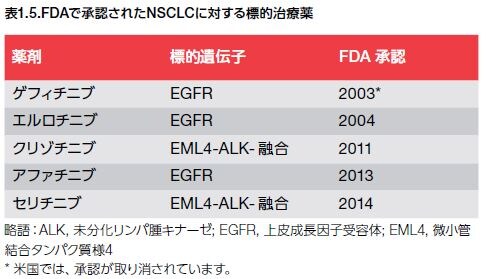
進行肺がん患者に対する治療は、現在、腫瘍学者によって標準化学療法レジメンに追加する治療オプションが拡大されています(表1.5)。食品医薬品局(FDA)および他の世界の医薬品規制当局は、適切な臨床ガイドラインおよび進行性肺がん患者の個々の診断に基づいて、NSCLCに対する複数の標的治療法を一次療法または二次療法として承認しました *42–45,47。
肺がんに対する承認済みのEGFR-および未分化リンパ腫キナーゼ(ALK)を標的とする治療については最適化が進められています。また、他の薬剤̶図1.9に示した他のドライバー変異の発がん促進活性を阻害すると考えられる低分子阻害剤および生物学的療法̶の探索が研究者によって継続されています *48,49。
異常なEGFRシグナル伝達は、NSCLCに関連するだけでなく、乳房、結腸、膵臓、頭頸部、および身体の他の部位の組織にも影響を与えます *50。
がんの分子プロファイリング
ドライバー変異
分子プロファイリング研究によって、さまざまながんの形態においてドライバー遺伝子が重複していることが明らかにされました(図1.11)。例えば、AKT1、PI3CAおよびPTENは特定の乳がんにおいて発現し、結腸直腸においても発現します。しかしながら、この図では、HER2は限局的な発現プロフィールとして乳癌の枠に表示されています(しかし、HER2は、特定のタイプの胃癌にも発現します)。同様に、BRAFおよびNRASの変異は、結腸直腸がんおよびメラノーマ(および他のタイプのがん)にも存在します。しかしながら、メラノーマには、乳がんや結腸がんでは頻繁には検出されないドライバー変異がいくつか存在します *51。
FDAで承認されている肺がんに対する標的治療薬の他に、乳、結腸、皮膚、腎臓、胃、および他の固形組織のがんに対する標的治療が承認されています。現在は特定の血液腫瘍に対する承認済みの標的治療の選択肢も存在しています *52。

 細胞周期およびDNA修復機構を調節する特定の遺伝子における変異は、異常増殖能をがん細胞に与え、正常細胞におけるアポトーシスを誘導する特定のタイプのDNA損傷の修復を妨げます。表1.2に表示されているいくつかの腫瘍抑制因子は、細胞周期調節、DNA修復機構、およびゲノム維持のための他の機構に必要とされます *53 。
細胞周期およびDNA修復機構を調節する特定の遺伝子における変異は、異常増殖能をがん細胞に与え、正常細胞におけるアポトーシスを誘導する特定のタイプのDNA損傷の修復を妨げます。表1.2に表示されているいくつかの腫瘍抑制因子は、細胞周期調節、DNA修復機構、およびゲノム維持のための他の機構に必要とされます *53 。
特性が決定されている特定のがんのタイプに関連するがん原遺伝子(および、それらのタンパク質産物)の例を以下に示します *54–57 。
- ALK(未分化リンパ腫キナーゼ; EML4と融合したALK)ー非小細胞肺がん
- GNA11(グアニンヌクレオチド結合タンパク質サブユニットα11)ー メラノーマ; 特に、ぶどう膜メラノーマ
- KRAS(カーステンラット肉腫ウイルスがん遺伝子ホモログ)ー結腸直腸がん、肺がん、膵がん、および他のがん
- c-KIT(チロシン-プロテインキナーゼ Kit)ーメラノーマならびに消化管、肺、他の組織および血液の特定のがん
- HER2(ヒト上皮成長因子受容体 2)ー乳房、消化管、および精巣の特定のがん
分子プロファイリングおよびがんサブタイプの分類によって新しい情報が提供され続けているのに伴い、既存の治療オプションを改良する方法を見出し、がんの進行を促進する細胞プロセスを妨げる新しい方法を発見するための試みが研究者によって続けられています。 

抗体: がん研究のための強力なツール
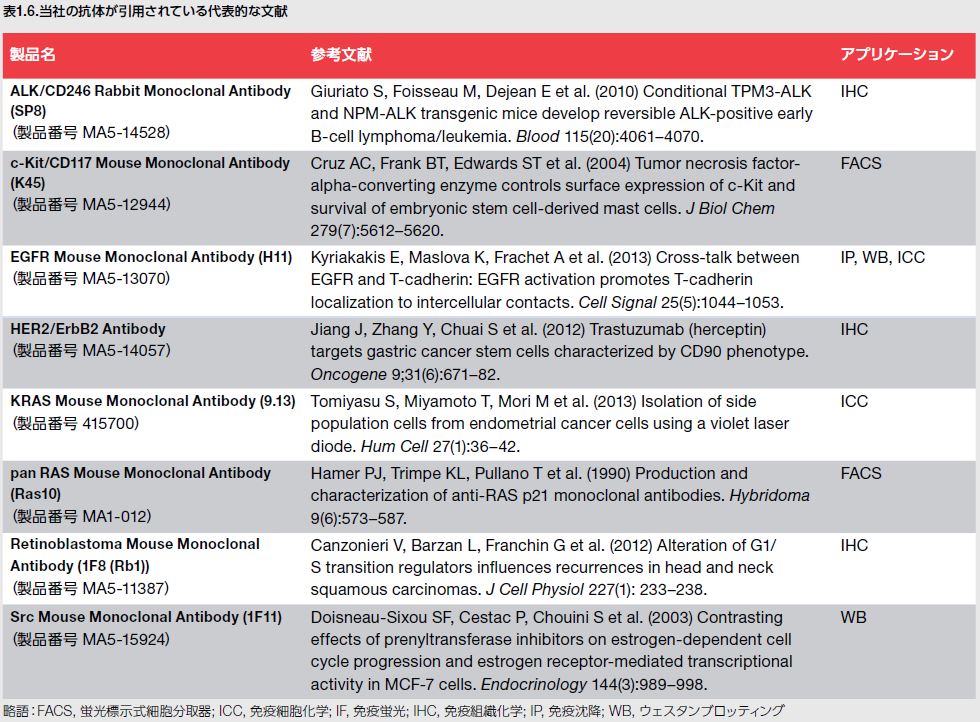
高品質な生物医学研究用抗体を提供する能力は、年を追うごとにがん生物学分野における新しい発見に貢献しています。当社では、数千ものがん研究用の一次および二次抗体を提供しています。お客様の研究のフォーカスが増殖シグナル伝達、転移、アポトーシス、オートファジー、代謝、炎症、腫瘍抑制因子、または他のどのようながん関連研究分野であっても、お客様の研究の成功を確実にする研究用抗体の幅広いラインナップを取り揃えています。当社の抗体が幅広いアプリケーションにおいて優れた実験結果の達成に寄与していることは、世界中の数千もの文献引用により実証されています。目的の抗体を見つけるためには、こちらの抗体検索ツールをご利用ください。
【無料ダウンロード】がん増殖シグナル伝達経路の探索ガイドブック
当記事は、がん増殖シグナル伝達経路の探索ガイドブックからの抜粋です。下記の内容を含むPDFは下記から無料でダウンロード頂けます。
~主な内容~
- がん概論
- がん増殖シグナル伝達
- PI3K/Akt/mTORシグナル伝達経路
- Ras/Raf/MEK/ERK(MAPK)シグナル伝達経路
- Wnt/β-カテニン シグナル伝達経路
- 抗体:がん研究のための協力なツール
【無料ダウンロード】がん研究を促進させる遺伝子解析技術ハンドブック
また、複数のがん研究領域における興味深い最新の発見を可能にする遺伝子解析技術を紹介するハンドブックも配布しています。併せてごらんください。
参考文献:
1. World Health Organization website (2015) Cancer key facts. http://www.who.int/mediacentre/factsheets/fs297/en/
2. American Cancer Society website (2014) Family cancer syndromes. http://www.cancer.org/cancer/cancercauses/geneticsandcancer/heredity-and-cancer
3. Anand P, Kunnumakkara AB, Sundaram C, et al. (2008) Cancer is a preventable disease that requires major lifestyle changes. Pharm Res 25(9):2097–2116. http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2515569/
4. Wroblewski LE, Peek RM Jr, Wilson KT (2010) Helicobactor pylori and gastric cancer: factors that modulate disease risk. Clin Microbiol Rev 23(4):713–739. http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2952980/
5. Di Lonardo A, Nasi S, Pulciani S (2015) Cancer: we should not forget the past. J Cancer 6(1):29–39. http://www.ncbi.nlm.nih.gov/pmc/articles/PMC4278912/figure/F2/
6. Frank SA (2007) Dynamics of Cancer: Incidence, Inheritance and Evolution. Princeton (NJ): Princeton University Press. pp 61–65. http://www.ncbi.nlm.nih.gov/books/NBK1569/
7. Marte B (2006) (1890) Cancer as a genetic disease. Nature Milestones: Cancer website. http://www.nature.com/milestones/milecancer/full/milecancer02.html
8. National Cancer Institute website (2014) Targeted therapies for cancer. http://www.cancer.gov/cancertopics/factsheet/Therapy/targeted
9. National Human Genome Research Institute website (2015) About NHGRI: a brief history and timeline. http://www.genome.gov/10001763
10. Nature Publishing Group (2006) Nature Milestones: Cancer, April. http://www.nature.com/milestones/milecancer/pdf/milecancer_all.pdf
11. Miller MJ, Foy KC, Kaumaya PT (2013) Cancer immunotherapy: present status, future perspective, and a new paradigm of peptide immunotherapeutics. Discov Med 15(82):166–176.
12. Stegmeier F, Warmuth M, Sellers WR, Dorsch M (2010) Targeted cancer therapies in the twenty-first century: lessons from imatinib. Clin Pharmacol Ther 87(5):543–552.
13. Naidoo J, Page DB, Wolchok JD (2014) Immune modulation for cancer therapy. Br J Cancer 111(12):2214–2219.
14. Fox Chase Cancer Center website (2015) Alfred G. Knudson Jr. summary page. http://www.fccc.edu/research/research-awards/knudson/index.html
15. Marte B (2006) (1953) Two-hit hypothesis. It takes (at least) two to tango. Nature Milestones: Cancer website. http://www.nature.com/milestones/milecancer/full/milecancer09.html
16. Knudson AG Jr (1971) Mutation and cancer: statistical study of retinoblastoma. Proc Natl Acad Sci USA 68(4):820–823.
17. Chial H (2008) Tumor suppressor (TS) genes and the two-hit hypothesis. Nature Education 1(1):177. http://www.nature.com/scitable/topicpage/Tumor-Suppressor-TS-Genes-and-the-Two-887
18. National Human Genome Research Institute website (1999) Scientists complete first chapter of book of life with decoding of first human chromosome. http://www.genome.gov/10002104
19. Weinberg R (2013) The Biology of Cancer, 2nd ed. New York (NY): Garland Science. p 269. https://books.google.com/books?id=MzMmAgAAQBAJ&pg=PA269&lpg=PA269&dq=Human+tumor+suppressor+genes+that+have+been+cloned&source=bl&ots=A0JkRa800f&sig=kKyxZPicikTAlDZL6ImeHvhiicI&hl=en&sa=X&ei=rZ-
1VPDMD4L9yQSb9oHACQ&ved=0CDIQ6AEwAjgK#v=onepage&q=Human%20tumor%20suppressor%20genes%20that%20have%20been%20cloned&f=false
20. The Medical Biochemistry Page website (2015) Introduction & table of familial cancer syndromes (noninclusive listing). http://themedicalbiochemistrypage.org/tumor-suppressors.php
21. Goodrich DW (2006) The retinoblastoma tumor-suppressor gene, the exception that proves the rule. Oncogene 25(38):5233–5243. http://www.nature.com/onc/journal/v25/n38/full/1209616a.html
22. Chin L, Hahn WC, Getz G, Meyerson M (2011) Making sense of cancer genomic data. Genes Dev 25(6):534–555. http://genesdev.cshlp.org/content/25/6/534.long
23. Hanahan D, Weinberg RA. (2000) The hallmarks of cancer. Cell 100(1):57–70.
24. Hanahan D, Weinberg RA. (2011) Hallmarks of cancer: the next generation. Cell 144(5):646–674.
25. Vogelstein B, Papadopoulos N, Velculescu VE, et al. (2013) Cancer genome landscapes. Science 339(6127):1546–1558.
26. Stratton MR, Campbell PJ, Futreal PA (2009) The cancer genome. Nature 458(7239):719–724. http://www.nature.com/nature/journal/v458/n7239/fig_tab/nature07943_F1.html
27. Heemskerk B, Kvistborg P, Schumacher TNM (2013) The cancer antigenome. EMBO J 32(2):194–203. http://onlinelibrary.wiley.com/doi/10.1038/emboj.2012.333/full
28. Bozic I, Antal T, Ohtsuki H, et al. (2010) Accumulation of driver and passenger mutations during tumor progression. Proc Natl Acad Sci USA 107(43):18545–18550. http://www.pnas.org/content/107/43/18545.abstract
29. Greenman C, Stephens P, Smith R, et al. (2007) Patterns of somatic mutations in human cancer genomes. Nature 446(7132):153–158.
30. Chen Y, Hao J, Jiang W, et al. (2013) Identifying potential cancer driver genes by genomic data integration. Sci Rep 3:3538. http://www.nature.com/srep/2013/131218/srep03538/full/srep03538.html
31. Lodish H, Berk A, Zipursky SL, et al. (2000) Section 24.2: Proto-Oncogenes and Tumor-Suppressor Genes. In: Molecular Cell Biology. 4th ed. New York (NY): W. H. Freeman and Company. http://www.ncbi.nlm.nih.
gov/books/NBK21662/
32. Martin GS (2004) The road to Src. Oncogene 23(48):7910–7917. http://www.nature.com/onc/journal/v23/n48/full/1208077a.html
33. Roskoski, R Jr. (2005) Src kinase regulation by phosphorylation and dephosphorylation. Biochem Biophys Res Commun 331(1):1-14.
34. Oláh E, (2005) Basic concepts of cancer: genomic determination. J Int Fed Clin Chem 16(2). http://www.ifcc.org/ifccfiles/docs/160206200501.pdf
35. De Roock W, De Vriendt V, Normanno N, et al. (2011) KRAS, BRAF, PIK3CA, and PTEN mutations: implications for targeted therapies in metastatic colorectal cancer. Lancet Oncol 12(6):594–603.
36. National Library of Medicine (US) website (2015) Genetics home reference. http://ghr.nlm.nih.gov/
37. Pao W, Hutchinson KE (2012) Chipping away at the lung cancer genome. Nat Med 18(3):349–351.
38. National Cancer Institute website (2014) Non-small cell lung cancer treatment (PDQ)®: general information about non-small cell lung cancer. http://www.cancer.gov/cancertopics/pdq/treatment/non-small-cell-lung/
Patient/page1#_124
39. American Lung Association website (2015) Lung cancer fact sheet. Mortality. http://www.lung.org/lungdisease/lung-cancer/resources/facts-figures/lung-cancer-fact-sheet.html
40. Reck M, Popat S, Reinmuth N, et al. (2014) Metastatic non-small-cell lung cancer (NSCLC): ESMO Clinical Practice Guidelines for diagnosis, treatment and follow-up. Ann Oncol 25 Suppl 3:iii27–39.
41. Chustecka Z (2013) EGFR inhibitors vs chemo in EGFR-positive NSCLC: no more trials. Medscape Medical News http://www.medscape.com/viewarticle/810379
42. Genentech (2014) Erlotinib. http://www.gene.com/download/pdf/tarceva_prescribing.pdf
43. National Cancer Institute website (2011) FDA approval for gefitinib. http://www.cancer.gov/cancertopics/druginfo/fda-gefitinib
44. National Cancer Institute website (2013) FDA approval for afatinib dimaleate. http://www.cancer.gov/cancertopics/druginfo/fda-afatinibdimaleate
45. Novartis (2014) Ceritinib. http://www.pharma.us.novartis.com/product/pi/pdf/zykadia.pdf
46. Normanno N, De Luca A, Bianco C, et al. (2006) Epidermal growth factor receptor (EGFR) signaling in cancer. Gene 366(1):2–16.
47. Rolfo C, Passiglia F, Ostrowski M, et al. (2015) Improvement in lung cancer outcomes with targeted therapies: an update for family physicians. J Am Board Fam Med 28(1):124–133. http://www.jabfm.org/
content/28/1/124.full.pdf+html
48. My Cancer Genome website (2015) Molecular profiling of lung cancer. http://www.mycancergenome.org/content/disease/lung-cancer/
49. Life Sciences Connect website (2014) Targeted therapy NSCLC part I: biologics. http://lsconnect. thomsonreuters.com/biologics-non-small-cell-lung-cancer-nsclc-part-current-status-2014/
50. Brand TM, Iida M, Li C, Wheeler DL (2011) The nuclear epidermal growth factor receptor signaling network and its role in cancer. Discov Med 12(66):419–432. http://www.discoverymedicine.com/Toni-MBrand/2011/11/22/the-nuclear-epidermal-growth-factor-receptor-signaling-network-and-its-role-in-cancer/
51. My Cancer Genome website (2014) Molecular profiling of melanoma. http://www.mycancergenome.org/content/disease/melanoma/ (see tabs for breast cancer, colorectal cancer, and melanoma)
52. National Cancer Institute website (2014) Targeted cancer therapies. http://www.cancer.gov/cancertopics/factsheet/Therapy/targeted
53. Hoeijmakers JH (2001) Genome maintenance mechanisms for preventing cancer. Nature 411(6835): 366–374.
54. UpToDate website (2015) Anaplastic lymphoma kinase (ALK) fusion oncogene positive non-small cell lung cancer. http://www.uptodate.com/contents/anaplastic-lymphoma-kinase-alk-fusion-oncogene-positive-nonsmall-
cell-lung-cancer
55. Chen X, Wu Q, Tan L, et al. Combined PKC and MEK inhibition in uveal melanoma with GNAQ and GNA11 mutations. Oncogene 33(39):4724–4734.
56. Eser S, Schnieke A, Schneider G, Saur D (2014) Oncogenic KRAS signalling in pancreatic cancer. Br JCancer 111(5):817–822.
57. Pittoni P, Piconese S, Tripodo C, Colombo MP (2011) Tumor-intrinsic and -extrinsic roles of c-Kit: mast cells as the primary off-target of tyrosine kinase inhibitors. Oncogene 30(7):757–769.
研究用にのみ使用できます。診断目的およびその手続き上での使用はできません。
![Rb [pSpT249/252] ABfinity™ Recombinant Rabbit Monoclonal Antibody](https://www.thermofisher.com/blog/wp-content/uploads/sites/13/2019/07/1-49.jpg)