今回は、長年に亘ってゴールドスタンダードとされてきた技術の基礎を、改めて振り返ってみましょう。すでにお察しの方もいるかと思いますが、そうです、キャピラリー電気泳動を使用したサンガーシーケンス(サンガー法)についてのお話です。
「なぜ、サンガーシーケンスと呼ばれているのか?」と、疑問に思っている方も多くいるかもしれません。サンガーシーケンスという名前は、今から40年以上前の70年代半ばに、この画期的な技術を開発したFrederic Sangerに由来しています。
では、早速サンガーシーケンスの原理を見ていきましょう。
※ブログ本文でYouTube動画内容をお伝えしていますので、合わせてご覧ください。
シーケンス反応の原理
すべては、短鎖のプライマーが、測定領域の隣に結合するところから始まります。4種類のヌクレオチドの存在下で、ポリメラーゼ酵素が、鋳型DNAの相補的なヌクレオチドをプライマーに結合させ、伸長させていきます。正確なDNA配列を決定するためには、この反応を途中で停止させ、伸長が停止した最後の塩基を特定する必要があるのですが、まずサンガーはリボヌクレオチドから酸素原子を取り除くことにより、この伸長反応の停止を実現しました。このようなヌクレオチドは、ジデオキシヌクレオチドと呼ばれ、これがリボヌクレオチドの代わりに取り込まれると、いわばレンチをギアに投げ込んで動きを止めるように、伸長反応を止める働きをします。つまり、ポリメラーゼ酵素はこの先DNA断片に、通常のヌクレオチドを追加することができなくなるのです。伸長反応を停止させ、その最後の塩基が何であるかを特定するため、ジデオキシヌクレオチドを各塩基4つの特定の色を標識し、識別できるようにしています。
このように、サンガーシーケンスでは、3’末端がジデオキシヌクレオチドで終わる、1塩基違いのさまざまな長さの反応産物が合成されます。
シーケンサーでの検出原理
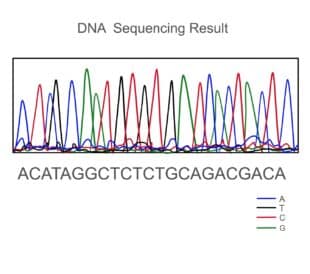
シーケンス反応で得られた、1塩基違いのさまざまな長さの反応産物を、シーケンサーを用いたキャピラリー電気泳動で分離させます。反応産物は、ゲルポリマーが充填された長いガラスキャピラリーに電気的に取り込まれ、DNA断片自体は負の電荷を帯びていますので、キャピラリー電気泳動中は、電界を印加することによって、陽極側に流れていきます。ゲルの中を移動する速度は、DNA断片の分子量に反比例し、短い断片は早く流れ、長い断片は時間がかかります。このプロセスで、1塩基違いの断片を分離していきます。キャピラリーの端には窓があり、そこにレーザーを当て、3’末端が蛍光で標識されたDNA断片が通過すると同時に励起します。励起された蛍光は、特異的な波長を放出しますので、それを光センサーで検出します。その後、ソフトウエアで検出された信号を読み取り、塩基情報に変換します。
4つのジデオキシヌクレオチド存在下でシーケンス反応を実行すると、最終的に末端が各塩基の蛍光で標識された1塩基違いのDNA断片が得られ、1塩基違いで分離、測定されます。そして、カラフルな波形で塩基配列が示されたデータファイルと、配列情報のテキストファイルが得られる、という流れです。
これがサンガーシーケンスの原理です。
まとめ
・サンガーシーケンスは、Frederic Sangerが開発した技術
・シーケンス反応では、途中で伸長反応を停止し、3’末端が各塩基を識別する蛍光で標識された、1塩基違いの断片を合成
・シーケンサーでのキャピラリー電気泳動で、1塩基違いの断片を分離し、流れてきた順に3’末端の蛍光を検出することで、塩基配列を決定
【無料公開中】サンガーシーケンスの基礎
このケミストリーガイドでは、ワークフローからアプリケーションまでサンガーシーケンスの基本原理を学べます。
研究用にのみ使用できます。診断用には使用いただけません。