▼もくじ [非表示]
シンプル、正確、お手頃なデジタルPCRによるNGSライブラリ定量
デジタルPCRは、NGSライブラリ定量をシンプルかつ正確に行い、マイクロチップ型電気泳動や分光光度計といった他の手法を用いたときに起こりがちな、ライブラリ濃度を誤って評価する可能性を軽減します。デジタルPCRは、あるライブラリにおいて、フォワードとリバースアダプターの両方をカバーするようにデザインされた特異性の高いアッセイを使用し、両方のライブラリアダプター配列を含む構造のみを検出します。
デジタルPCRは標準曲線やリファレンスサンプルを使用せず、NGSライブラリを正確に定量できるので、実験の手間や失敗のリスクを最小限に抑え、シーケンシングワークフローにかかるコストを軽減できます。
また、デジタルPCRは、シンプルなワークフローにより、わずか数時間でライブラリ濃度を精密に測定できます。そのため、ランニングのやり直しやサンプル調製の失敗のリスクを最小限に抑えます。NGSライブラリの濃度は、高すぎても低すぎても、トータルのリード数の減少、データ収率やスループットの低下につながります。
どのようなときにデジタルPCRをNGSライブラリの定量に使用するか
標準的なライブラリには増幅ステップが必要です。これにより正しく作製されたライブラリ(AおよびBアダプターの両方の配列を含むライブラリ)が濃縮されます。増幅ステップは、緑の楕円で示すようなAあるいはBアダプターの片方しか持たない無効なライブラリの生成を最小限に抑えます。無効なライブラリと有効なライブラリが混在する中で、分光光度計などでライブラリ濃度の定量を行うことは不十分といえます。
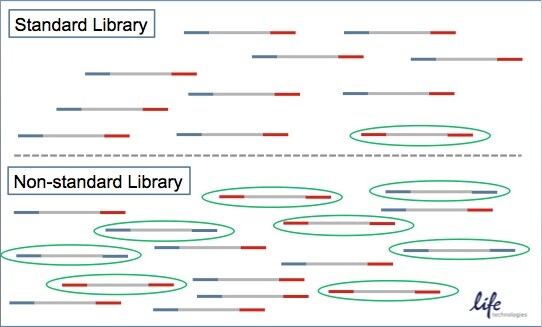
デジタルPCRは、特にPCRステップを含まないプロトコール(Non-Standard Library)で作製したライブラリの定量に特に有効です。Non-Standard Libraryで作製すると、緑の楕円で示されているような、無効なライブラリの割合がより高くなります。
Non-Standard Libraryのプロトコールで作製されたライブラリにおいて、増幅ステップは、正しく作製されたライブラリの濃縮に有用です。現在、NGSライブラリの定量に使用されている方法としては、分光光度法やqPCR法などがあります。
分光光度法は、長い間DNAやRNAの定量に使用されてきました。しかし、紫外線吸光度法による測定は特異的ではなく、DNA、RNA、タンパク質の間の区別ができません。また測定値は、遊離ヌクレオチド、塩、エタノールやフェノールなどの有機物や塩基組成のばらつきなどに影響を受け、実際の濃度より高い濃度が示される場合があります。分光光度計の感度は、低濃度のDNAやRNA定量において多くの場合不十分です。
リアルタイムPCR は、NGSのライブラリ定量を行う有用な方法ですが、リファレンスサンプルや標準曲線の作成が必要です。スループットが求められる研究室で、異なる種類のライブラリを並行してシーケンスを行う場合、複数の標準曲線を作成する必要がありますが、作業が煩雑になり、コストや所要時間が増え、エラーの可能性を高める可能性があります。
1つの大きな潜在的エラーとしては、PCRバイアスがあります。これは、テンプレートの増幅効率の差やPCRの後半に生じる増幅産物による阻害です。多くのプロトコールでは、PCRバイアスを避けるために増幅を行わない傾向にあり、これが正しく作製されるライブラリの割合の低下につながっています。
特に、分光光度計は、正しく作製されたライブラリと正しく作製されなかったライブラリの両方を検出するため、ライブラリ濃度が過大評価される可能性があります。
Applied biosystems™ QuantStudio™ 3D デジタルPCRシステムを、Ion Torrent™、あるいはIllumina® ライブラリ定量用のTaqMan™ アッセイとともにご利用いいただくと、正しく作製されたライブラリのみを検出できるので、濃度の過大評価の問題を軽減し、精密なライブラリ定量が可能です。正確・精密なNGSライブラリの定量は、シーケンシングデータの収率を最大化するためにも、Ion Torrent™や他のNGSワークフローにおいても重要なステップです。
QuantStudio™ 3Dを用いた Ion Torrent™ NGSライブラリの精密な定量
Ion Torrent プラットフォームなどを用いたNGSワークフローの中で、テンプレート調製ステップは、シーケンシングの収率を最適化するために重要です。このステップはライブラリのインプット量に大きく左右されます。ライブラリ濃度が高すぎても低すぎても、トータルのリード数およびスループットにマイナスの影響を及ぼします。
QuantStudio 3D デジタルPCRシステムでは、PCR反応液(希釈したサンプル+ QuantStudio 3D dPCR Master Mix + TaqManアッセイ)を、20,000個のウェルを持つチップに分配し密閉します。ターゲット分子の1コピーが1ウェルに含むか含まないように、サンプルを最初に希釈し、デュアルフラットブロックのサーマルサイクラ―で増幅を行います。ターゲット分子が含むウェルはポジティブ反応、含まないウェルはネガティブ反応となり、ネガティブ反応のウェルの割合を用いてサンプル中のターゲット分子を定量します。
絶対定量: 最終的なライブラリ濃度を求める前に、ライブラリサンプルの希釈率と反応液の調合時(チップ上での)の希釈率に基づいてデータを調整しなければなりません。 このプロセスは、QuantStudio™ 3D AnalysisSuite™ Cloud ソフトウェアをご利用いいただけます。
相関: Ion OneTouch 2 システムでテンプレートビーズを調製するためには、濃度計算値 (nM) に基づいてライブラリを希釈する必要があります。エンリッチメントを行う前に、Qubit® 3.0 Fluorometerを使用して、サンプル中で生成されたテンプレートビーズの品質を評価するために品質管理のステップが必要です。ビーズの10~30%が増幅テンプレートと結合するようにライブラリを希釈したときに、最適なライブラリ定量ができます。この推奨範囲内に入るサンプルからは、おおむね質の高いデータが得られます。
まとめ
最適なNGSデータ収率を得るには、質の高いテンプレート調製が必要です。デジタルPCRでライブラリ定量を行うことで、シーケンシングにかかる全体的なコストの削減につながります。それは、ダウンストリームでの追加作業やステップの繰り返しを回避し、シーケンスの品質を確保できるからです。QuantStudio 3DデジタルPCRシステムとライブラリに特異的なTaqManアッセイは、サンプルの取扱いにかかる手間を最小限に抑え、標準曲線やリファレンスサンプルを使用することなく、高精度のテンプレート調製を実現した、お手頃で使い易いデジタルPCR法を提供します。
研究用にのみ使用できます。診断用には使用いただけません。






