前回に引き続き、ゲノム編集の初心者Aさんによる「初めてのゲノム編集実験」シリーズ、第7回です。
Aさんはこれまで、ターゲットとなる遺伝子の機能を欠失させるノックアウト法について学んできました。しかしゲノム編集には、目的の遺伝子配列を挿入するノックイン法もあります。
第7回では、このノックイン法の実験デザインやポイントについてご紹介しましょう。
▼もくじ [非表示]
ゲノム編集のノックインとは?
Aさん:
今までゲノム編集のノックアウトについて学んできましたが、今後のために教えてください。目的部位に特定の配列を挿入するとき、どういう流れで実験をするんでしょうか。
先輩:
ノックインのことだね。基本の流れは同じだよ。ゲノム編集の主役2人のほかにドナー核酸が必要になるんだ。
Aさん:
ゲノム編集の2人の主役、Cas9とガイドRNAですよね!これに加えてドナー核酸が必要…。ドナー核酸ってなんでしょうか。
先輩:
ドナー核酸は、ゲノム編集で挿入したい配列を含んだ核酸だよ。ベクターや短い一本鎖オリゴがよく使用されるね。
挿入したい配列(SNPや蛍光タンパク質の遺伝子など)が決まっている場合は、ノックインでゲノム編集を実施します。ノックインシステムは、ランダム変異(ノックアウト)の実験と異なり、挿入する配列を含んだドナーベクターや一本鎖オリゴ(single strand oligo)の準備が必要です。まずは図1でノックインの仕組みを確認しておきましょう。

図1:ノックインの仕組み
ノックインシステムでは、まずノックアウトと同様に、ガイドRNAとCas9によってターゲット部位に二本鎖DNA切断(Double Strand Break:DSB)が起こります。このとき、ドナー核酸(ドナーベクターや一本鎖オリゴなど)が細胞内に共存すると、DSB後に細胞内の相同組み換え(Homology directed repair:HDR)が起こり、ドナー配列または一本鎖オリゴの配列がゲノムに取り込まれ、ノックインが起こるという仕組みです。
Aさん:
ドナー核酸はどんなデザインでもいいんですか?SNPを入れ替えたいときは1塩基だけ入れる…ってことはないですよね。
先輩:
ないね。ドナー核酸には挿入したい配列と、DSBと隣接した配列と相同性を持つアーム配列の両方が必要だよ。そして、「相同組み換えが起こりやすいデザイン」っていうのがあるんだ。
一般的によく使われるドナー核酸として、ベクターと一本鎖オリゴがあります。使い分けは、数塩基や数十塩基の変異など、挿入したい配列や入れ替えたい配列が短い場合は一本鎖オリゴDNA、それ以上の長い配列はドナーベクターを用いることが多いです。
どういうデザインが相同組み換えを起こしやすくするのかなど、それぞれのポイントを下記にまとめました。
<ドナーベクター、デザインと選択のポイント>
- 長い配列の場合はドナーベクターが推奨
- 挿入したい配列の左右に、ターゲットの周辺配列と相同的なアーム配列(500 bp以上)を設ける
- 組み換え率は相同領域長に依存し、一般的に挿入配列が長いほどアーム(相同配列)も長く必要
- DSBから少なくとも100 bp以内から相同領域を取得
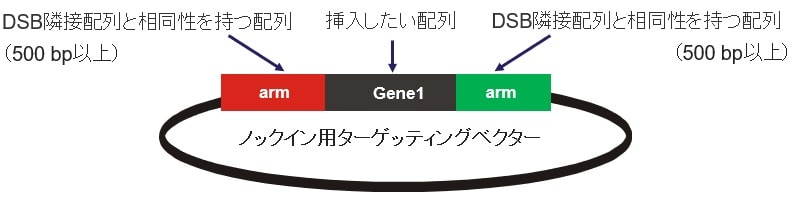
図2:ドナーベクターのデザイン
<一本鎖オリゴDNA、デザインのポイント>
- 数十塩基など短い配列の場合に推奨
- 挿入したい配列の左右に、ターゲットの周辺配列と相同的なアーム配列(40~90塩基)を設ける
- アーム配列は長い方が比較的ノックイン効率が良い
- センス、アンチセンス鎖どちらでも対応可能
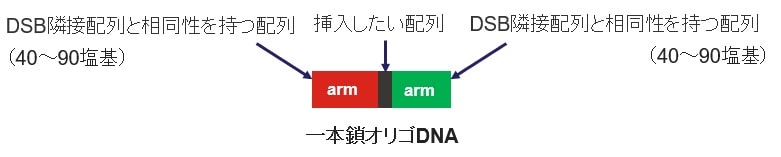
図3:ドナー一本鎖オリゴDNAのデザイン
Aさん:
一本鎖オリゴDNAは合成で手に入れられますよね。精製グレードはどのくらいがいいんですか?あと、ベクターはどうやって作製すればいいんでしょう。
先輩:
一本鎖オリゴDNAの精製度はPAGE精製が推奨だよ。ベクターは、どういった方法でもいいけど、Invitrogen™ GeneArt™ Seamless kitなら、3断片をベクターに導入できるよ。
あとは、Invitrogen™ TrueTag™ Donor DNA Kitシリーズみたいなキットを使えば、GFPやRFP、HA、6×His-tagなどが挿入できるドナー核酸が数時間で作れるよ。蛍光色素やおなじみの配列を入れたい場合は、こういう製品を探してみるのも手だね。
ノックインのガイドRNAデザイン
Aさん:
ところでノックインのガイドRNAとCas9は、ノックアウトと同じものでよいんでしょうか。
先輩:
Cas9は同じでいいけど、ガイドRNAは注意が必要だよ。
ノックインの場合は、二本鎖の切断(DSB)が起こった部位からノックイン部位までの距離が10塩基以内になるようにデザインすると、相同組み換えの効率が高いことがわかってるんだ。
Aさん:
10塩基以内ですか?PAM配列もあるし、デザインが限定されそうですね。
先輩:
そうだね。でも、ノックイン用のガイドRNAもソフトウエアでデザインできるから、任せちゃえばいいよ。
ノックイン用のガイドRNAは、第2回でノックアウト用のガイドRNAデザインツールとしてご紹介した「TrueDesign Genome Editor」を用いてデザイン可能です。
まずは第2回にて解説した方法に従って、「TrueDesign Genome Editor」を開き、1. Select a gene editing experimentをクリックしましょう。ノックアウトのときは「Gene Knockout」を選択しましたが、ノックインのときは、その右横の5つの選択肢の中から選びます。
それぞれの選択肢の意味は、以下のとおりです。
- Gene Knockout:ノックアウト実験
- Gene Tagging:ノックイン実験のうち、GFPやRFPなど蛍光タンパク質を挿入する場合
- Insertion:ノックイン実験のうち、30塩基までの特定配列を入れる場合
- Deletion:ノックイン実験のうち、30塩基までの特定配列を除去する場合
- SNP Edit:ノックイン実験のうち、SNPの1塩基を変更したい場合
- Replacement:ノックイン実験のうち、30塩基までの特定配列を、別の特定配列に置き換える場合
ここでは一例として、「SNP Edit」を選んで続けます。
「2. Select Species」で生物種を選択します。ここに表示されている以外の生物種はデザインができませんのでご注意ください。
一例として「Human」を選択し、「Next」をクリックします。
「3. Select Input Type」で、遺伝子の検索の仕方を選びます。遺伝子名から検索したいときは「Gene Symbol」、配列のときは「DNA Sequence」、染色体のロケーションから検索するときは「Chromosome Locus」を選んでください。
ここでは、多くの人が選ばれるであろう「Gene Symbol」を選択します。
Gene Symbolとして、試しに「KLF4」と入れてみると、選択肢が表示されました。
近い遺伝子名がある場合、このように選択肢が表示されます。ノックインしたい遺伝子を選び、「Search Gene」をクリックしましょう。ここでは上の選択肢を選びます。
「5. Select one of the following xx transcript(s)」では、その遺伝子にバリアントがある場合、いずれかを選ばなくてはなりません。「Transcript ID」をクリックすると、NCBIのページに飛んで、詳細が確認できます。
一例として、上の遺伝子を選びます。Selectにチェックを入れ、「Next」をクリックしてください。
下図のような画面が表示されます。まずは上半分からご説明しましょう。
一部拡大すると、下図のようになります。
最上段の図は遺伝子マップで、Intron、Exon、UTR、Flanking sequenceに分かれています。それぞれの色分けは下記のとおりです。![]()
上から2段目の図「Sequence Editor」は、遺伝子マップの青い四角内を拡大した図で、遺伝子配列、アミノ酸配列が書かれています。
では、この遺伝子に対してSNP Editできるノックイン用のガイドRNAをデザインしてみましょう。まずは「Sequence Editor」で、置換したいSNP、つまり1塩基をクリックしてください。そうすると、画面下部に、どの塩基に置換するか選択肢が表示されます。
1塩基選んで「OK」をクリックすると、下図のように「Edit List」に内容が入ります。
「Next」をクリックすると、ガイドRNAのデザインが始まります。
3種類のガイドRNAがデザインされました。それぞれの項目について説明します。
- CRISPR Target Sequence:
ガイドRNA配列です。クリックすると、ページ上のSequence Editor内で該当配列がハイライトされます。 - PAM Site:
SqCas9のPAM配列です。
※このソフトウエアではSqCas9の配列しかデザインができませんのでご注意ください。 - Score(%):
アルゴリズムの合致率を示すスコアです。合致率が高いほどスコアが高くなりますが、スコアが高いからと言って活性が高いとは限りません。目安のひとつとお考えください。 - Off-Targets:
オフターゲットを引き起こす可能性のある配列の数です。クリックすると、各オフターゲットの配列が確認できます。 - Edit site distance:
二本鎖切断が起こった箇所からSNPまでの距離です。10塩基以内にする必要があります。 - Donor DNA:
ドナーDNAの配列です。変異やアームとなる相同配列を含んだ状態で表示されています。
Target Sequenceの右横についている緑色のチェックは、表示された4種類の中で推奨される配列です。緑色のチェックがついている配列の中に優劣はありません。この例では3つの配列にチェックがついていますが、3つの配列が同じくらい推奨されることを示しています。
ゲノム編集では、ガイドRNAはまず1種類を購入して試すことが多いです。注文したいガイドRNAのいずれかひとつにチェックを入れて、Nextをクリックしてください。
注文ページに飛びます。
このページの項目では、注文するものにチェックを入れる必要があります。各項目について説明しましょう。
<左列>
- CRISPR gRNA Format:チェックを入れてください。デザインしたガイドRNAをオーダーできます。容量は1.5 nmolのみです。
- Cas9 Format:チェックを外してください。代理店からご注文をお願いいたします。
- Cas9 Reagent:チェックを外してください。代理店からご注文をお願いいたします。/li>
- Transfection Reagent:チェックを外してください。代理店からご注文をお願いいたします。
※「Cas9 Format」「Cas9 Reagent」「Transfection Reagent」は、日本ではここから注文ができません。もしチェックをしたままCartに入れると、「注文できません」というアラートが出ますので、ここで外しておいてください。
<右列>
- Primers:チェックを入れると、ミスマッチ特異的ヌクレアーゼ切断解析(第5回参照)を実施するときのPCR用プライマーを注文できます。容量は25 nmolのみです。
「View」をクリックすると、ガイドRNAに対してデザインされたForward PrimerとReverse Primerの配列が確認できます。デフォルトだと3つすべてにチェックが入っているので、1種類でよい場合は、どれか1つだけチェックを入れておきましょう。
※オフターゲット配列に対するPrimerはこのサイトでは注文できません。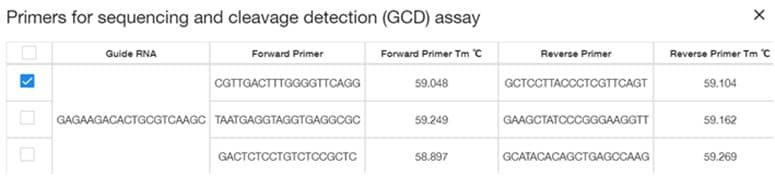
- Other products You May Need:すべてチェックを外してください。その他の試薬が必要な場合は、別途代理店からご注文をお願いいたします。
- Download Designs and Protocol:クリックし、ご注文内容のダウンロードと保存をおすすめします。サイトでデザインしたオフターゲット配列なども保存しておけるためです。
チェックを入れ終えたら、「Add to Cart」をクリックしてカートに入れましょう。
このようにデザインされたcrRNAを用いてゲノム編集を行うことで、オフターゲット効果を可能な限り防ぐことが可能です。しかし、オフターゲットがゼロになるわけではありません。そのため、ゲノム編集株を樹立した際は、オフターゲットによる変異を確認しましょう。このツールの「Off-Targets」で表示された配列付近がオフターゲットを引き起こす可能性の高い配列ですので、その付近について、シーケンスなどでオフターゲットの変異が導入されていないことを確認する必要があります。
Aさん:
デザインしたガイドRNAとCas9とドナー核酸をトランスフェクションした後の流れは、ノックアウトと同じですよね? ノックインで何かほかに気を付けるポイントはありますか?
先輩:
そうだね、ひとつは、根気よく行うことかな。相同配列の効率は高くないから、低い効率であることを踏まえてクローニングの数を増やすとか、工夫するといいよ。
ふたつめは、ノックインにこだわらないこと。ノックインは、「目的の場所」に「挿入したい遺伝子や塩基がある」、この2つがそろったときに選択する方法だから、たとえば挿入したい遺伝子や塩基がどこに入ってもいい場合は別の方法もあるよ。状況に応じて実験手法を選ぶことも、実験成功のポイントのひとつだね。
まとめ:ノックイン用のドナー核酸とガイドRNAのポイント
- ノックインのときはCas9、ガイドRNAのほか、ドナー核酸が必要
- ドナー核酸で主に使われるのはベクターと一本鎖オリゴDNA。入れたい挿入配列の長さによって決めましょう。
- ノックイン用ガイドRNAは、DSB部位からノックイン部位までの距離が10塩基以内になるようにする
- ノックイン用ガイドRNAは、デザインツールでデザインが可能
さて、計7回に亘って連載してきた「そういうことだったのか!ゲノム編集実験」シリーズは本ブログで最終回です。読者の皆さまのゲノム編集実験がつつがなく進みますように。もしご実験で迷うことがあれば、サーモフィッシャーサイエンティフィックまでお気軽にお問い合わせください。
<ゲノム編集実験シリーズ>
そういうことだったのか ! ゲノム編集実験(CRISPR/Cas9) ~第1回 CRISPR/Cas9システムの原理~
そういうことだったのか ! ゲノム編集実験(CRISPR/Cas9) ~第2回 ガイドRNAのデザイン~
そういうことだったのか ! ゲノム編集実験(CRISPR/Cas9) ~第3回 3つのCRIPSR/Cas9実験系~
そういうことだったのか ! ゲノム編集実験(CRISPR/Cas9) ~第4回 トランスフェクション~
そういうことだったのか ! ゲノム編集実験(CRISPR/Cas9) ~第5回 変異の確認方法~
そういうことだったのか ! ゲノム編集実験(CRISPR/Cas9) ~第6回 オフターゲット効果~
そういうことだったのか ! ゲノム編集実験(CRISPR/Cas9) ~第7回 ノックイン~
<ゲノム編集の基礎的な内容>
ゲノム編集の原理と手引き

CRISPR/Cas9ゲノム編集を始める方におすすめ!トレーニングと試薬がセットになったスターターパック
技術革新が著しいCRISPER/Casゲノム編集分野において、試薬の選定に戸惑う方も少なくありません。またgRNA(ガイドRNA)のデザイン方法やトランスフェクションのノウハウは実験結果を左右する重要なポイントです。
そこで講義、実技トレーニング、必要な試薬をセットにしたとてもお得なパッケージを準備しました。試薬の選定からトラブルシュートまで、エキスパートがあなたの実験系の立ち上げを全力でサポートします。
[無料公開中]CRISPRゲノム編集リソースガイド
ゲノム編集の基礎の復習、より深い知識の獲得、または最新の研究ツールの検索ができます。ゲノム編集の中核として、初心者から経験者まで、豊富で信頼できる技術的な内容を提供しています。
研究用にのみ使用できます。診断目的およびその手続き上での使用はできません。