▼もくじ [非表示]
リアルタイムPCRにおける核酸精製法
リアルタイムPCRに用いる核酸の精製を行う前に、サンプルの種類(細胞または組織)および適切な精製方法の選択について考察することが必要です。DNAおよびRNA分離法により、簡便性や、有機溶媒の必要の有無、および回収した核酸の純度(DNA(RNA精製の場合)、タンパク質、有機溶媒の残存)が異なります。
今回は、リアルタイムPCRにおける核酸の精製および定量方法について、主としてRNA分離についてご紹介しますが、ガイドラインのほとんどはDNAの分離にも当てはまります。
有機溶媒を用いた精製法
1-Step の試薬ベースの有機抽出法は、RNAを様々な種類の細胞および組織から精製するのに非常に効果的な方法です。多くのプロトコルでは、核酸をRNaseの作用から保護し、核酸の完全性を維持しながら細胞を破壊して細胞成分を溶解するために、フェノールとグアニジンイソチオシアネートの混合物を使用します。通常、そこにクロロホルムを添加し、遠心分離によって混合物を水相と有機相に分離します。グアニジンイソチオシアネートの存在下ではRNAは水相に存在し、DNAおよびタンパク質は有機相および中間相に移動します。その後RNAをイソプロパノール沈殿により水相から回収します。
この方法は比較的迅速であり、高収量でRNAを得ることが可能ですが、毒性のある化学薬品を使用することが必要で、他の方法と比較してより高くDNAが残存する可能性があります。残留するグアニジン、フェノールまたはアルコールがcDNA合成効率を著しく低下させる可能性もあります。
シリカビーズまたはフィルターベースを用いた精製法
ほとんどのシリカビーズまたはフィルターベースの方法では、RNAを内在性のRNaseの作用から保護するカオトロピック塩であるグアニジンイソチオシアネートの存在下で試料の溶解および均質化を行います( Biochemistry 18:5294 (1979))。均質化の後、試料にエタノールを添加してRNAをシリカベースのビーズまたはフィルターに結合させ、不純物を洗浄によって効率よく除去します(Proc Natl Acad Sci USA 76:615(1979))。精製したTotal RNAは水で溶出します。
この方法は有機溶媒抽出法よりもさらに迅速で、フェノールを必要としません。RNAの収量は溶媒抽出ほど高くありませんが、タンパク質、脂質、多糖類、DNAおよび精製試薬に関する純度は通常有機溶媒抽出よりも良好です。洗浄が不完全だとグアニジンおよびエタノールが残存し、cDNA合成効率にともに悪影響を与える可能性があります。
RNAの品質の評価
RNAの品質と量の評価においては、配慮すべき重要点がいくつかあります。A260/A280 比が1.8から2.0 の間であることを確認してください。比が1.8 以下である場合にはタンパク質の汚染が示唆され、反応効率を低下させる可能性があります。A260/A230 比は、カオトロピック塩であるグアニジンイソチオシアナートおよびフェノールなど、酵素反応を阻害する、フェノール環を有する成分の残存の評価に有用です。RNAの分解度を、変性ゲル上またはAgilent Bioanalyzerなどの装置上で評価してください(図)。
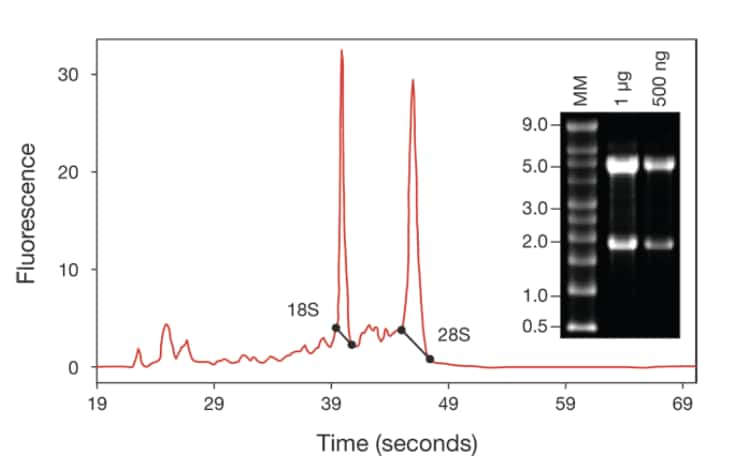
図 RNAの分解度を示す、Agilent Bioanalyzerトレースおよびゲルイメージ。2つの蛍光ピークは18Sおよび28SのrRNAバンドに相当します。分解していない哺乳類のtotal RNA は、18S および28S のrRNAを表す2 本のバンドまたはピークを示します。一般的に、28S rRNA の輝度は18S rRNA の2 倍です(あるいはAgilent Bioanalyzerトレースにおけるピーク下面積が2倍です)。
Agilent Bioanalyzerでは、RNAの品質決定をさらに進化させて、RIN(RNA integrity number)値を算出します。RIN値は、分解産物も含めた全体のトレースから算出され、通常rRNAピークのみを評価するよりも優れています。
異なる組織由来のRNAに関してRIN値を比較することにより、品質の標準化および一貫性の維持が可能です。
定量の精度
RNAの定量のためには、より高い感度、精度および高スループットを実現するようにデザインされた、Invitrogen™ RiboGreen™ 色素およびInvitrogen™ PicoGreen™ 色素などの蛍光色素を用いる方法が、UV吸収法よりも優れています。UV吸収測定法では核酸と遊離のヌクレオチドを識別することが不可能です。実際、遊離のヌクレオチドは260nmにおいて核酸よりも吸収が高くなります。同様に、UV吸収測定法では同一試料においてRNAとDNAを識別できません。さらに精製した核酸に含まれることの多い夾雑物はUV吸収を示します。最後に、UV吸光光度計は測定のためにかなりの量の試料を消費します。蛍光色素に関しては幅広い種類の色素が入手可能ですので、核酸を遊離のヌクレオチドから識別することが可能な色素、同一試料においてDNAをRNAから識別することが可能な色素および一般的な夾雑物に感受性のない色素など、前述のすべての欠点を克服することのできる試薬を選択することが可能です。Invitrogen™ Qubit™ Quantitation Platform は、DNA、RNAまたはタンパク質に結合すると蛍光性になる最先端の蛍光色素を用いる、Quant-iT 蛍光技術を使用しています。Invitrogen™ Quant-iT™ Assay Kit は研究対象の分子(夾雑物を含まない)の濃度のみを測定できるため、特異性が高く、UV吸収測定よりも正確な結果が得られます。しかも、一般的に蛍光色素を使用する定量法は非常に感度が高く、少量の試料しか必要としません。
遺伝子発現研究におけるゲノムDNAの残存
前回の記事において、プライマーデザインが、リアルタイムPCR反応において残存DNA由来の増幅を排除するための第一歩であるということをご紹介しました。RNAの精製段階における試料のDNase処理はDNAをもとからコントロールすることを可能とする方法です。従来のDNase I 酵素に加えて、当社では、触媒的に野生型DNase Iよりも優れた、高活性Invitrogen™ TURBO™ DNase を提供しています。TURBO DNaseは、RT-PCRの障害となるごく少量のDNAも除去することが可能です。DNase処理は、精製方法に応じて、溶液中でもカラム上でも行うことができます。カラム上でのDNase処理にはシリカマトリックス抽出を使用するのが一般的で、塩洗浄が酵素自身を除去するため、溶液中の処理とは異なり、EDTAの存在下で加熱による不活化を行う必要はありません。欠点としては、カラム上での反応にはより大量の酵素が必要です。
溶液中でのDNase反応は、従来65°CにおけるDNaseの加熱不活化を必要とします。反応に必要なマグネシウムが、この温度においてはマグネシウム依存性のRNA加水分解を引き起こす可能性があります。Invitrogen™ DNA-free™ キットおよびInvitrogen™ TURBO DNA-free™ キットは、新規のDNase不活性化試薬を使用してこれらの問題を回避します。DNase酵素を反応から除去するのに加えて、不活性化試薬は二価の陽イオンに結合し反応緩衝液から除去します。このため反応効率に影響を与える可能性のある二価の陽イオンをRT-PCR反応に使用することへの恐れを抑えることができます。
【無料公開中】リアルタイムPCRハンドブック
このハンドブックでは、リアルタイムPCRの理論や実験デザインの設計など、リアルタイムPCRの基礎知識が掲載されています。リアルタイムPCRを始めたばかりの方やこれから実験を考えている方にうってつけのハンドブックです。PDFファイルのダウンロードをご希望の方は、下記ボタンよりお申し込みください。
研究用にのみ使用できます。診断用には使用いただけません。


