遺伝子発現解析は、サンプル間での遺伝子発現レベルをタンパク質もしくはRNAを用いて比較する研究手法です。これらの手法では表現型の違いを遺伝子レベルで解明することができ、さらに詳細な知見を得るのに役立つため欠かすことのできない技術といえます。
遺伝子発現解析では主にqPCRや次世代シーケンスによるRNA-Seqが用いられていますが、それぞれ一長一短あります(表1)。qPCRでは1遺伝子につき通常1カ所にプライマーを設計し発現変動をモニターします。非常に精度が高い、迅速に結果が得られるという利点がありますが、同時に100以上の遺伝子を解析する際や、多サンプルを対象とする際に実験的な手間がかかることが難点です。また解析対象遺伝子数に比例してRNA量が多く必要になるため、サンプル量が限られる場合に実験デザインついて考慮が必要となります。

表1:各種アプリケーションの特徴
NGSによるRNA-Seqの場合には全転写産物を網羅的に解析できるという大きな利点がありますが、qPCRよりも実験コストが高額になる上に、着目したい遺伝子以外の解析結果まで得られることから、実験目的によっては情報過多となることがあります。また発現量の多い遺伝子に大部分のリードがマッピングされてしまうため、低発現の遺伝子の発現量を正確にモニターできないといった技術的課題も考慮する必要があるなど、解析にはそれなりの経験が要求され、qPCRと比べ敷居が高いことは否めません。
今回、Ion AmpliSeq™ RNAと呼ばれる、特定の遺伝子にターゲットを絞ったNGS遺伝子発現解析アプリケーションをご紹介いたします(表1)。この手法は10 ng程度と少ないサンプル必要量で12~1,200までの着目したい遺伝子を1チューブで同時に解析でき、貴重なサンプルでも網羅的な解析の道を開くことが可能です。
▼こんな方におすすめです!
・NGSを用いた発現解析に興味がある
・サンプルやターゲット遺伝子数が多く、qPCRでの実験だと規模が大きすぎる
・目的遺伝子が低発現のため、RNA-Seqでは正確な解析ができない
▼もくじ [非表示]
Ion AmpliSeq RNAテクノロジー -着目したい遺伝子だけに絞り込んだ発現解析-
NGSでの遺伝子発現解析手法として広く用いられているRNA-Seqは転写産物全長を解析対象としています(図1左)。解析の大まかな流れとしては、まずTotal RNAもしくはmRNAを断片化したものを逆転写した後、cDNAをNGSで解析可能な形に加工(ライブラリー化)し、得られたシーケンスリードを遺伝子のエクソン全体にマッピングしていきます。この結果から遺伝子ごとにいくつのリードがマッピングされたかの情報を取得し、統計処理などを駆使して解析を行います。
一方、Ion AmpliSeq RNAテクノロジーでは、興味のある遺伝子につき1カ所にプライマーを設計しますのでqPCRに近い考え方です。複数の対象遺伝子に対するプライマーペアを1本のチューブにまとめたパネルと呼ばれるプライマーセットを準備し、RNAを逆転写して得られたcDNAを鋳型に1チューブ内で複数ターゲットのPCR増幅(マルチプレックスPCR)を行い、シーケンス解析を行います(図1右)。
各遺伝子の転写産物の量が反映された状態でシーケンスが行われるため、取得リード数からサンプル間の遺伝子発現量の変化を見積もることができます。

図1:NGSを用いた発現解析手法
Ion AmpliSeq RNAテクノロジーを用いた発現解析アプリケーションとしては、約20,000遺伝子を対象にしたIon AmpliSeq™ Transcriptome Human/Mouse Gene Expression Kit(A26325, A36553)と、12~1,200のヒト遺伝子を任意に選択可能なIon AmpliSeq™ RNAカスタムパネルが提供されており、網羅的な解析から、ターゲットを絞った解析まで対応可能です(表1)。Ion AmpliSeq RNAテクノロジーでは、興味のある1遺伝子につき1カ所に検出部位を絞ったプライマーでライブラリー調製を行います。RNA-Seqのように全エクソンをカバーする必要がないため、リード数を絞った発現解析を行うことができるのが大きな利点で、RNA-Seqよりコンパクトな解析が可能です。また、Ion AmpliSeq RNAカスタムパネルでは任意の遺伝子のみを選択できるので、低発現な遺伝子が対象となる際に問題となりやすい、発現量の多い遺伝子の持つエクソンに取得リードの大部分を占領されてしまう現象は心配する必要がありません。Ion AmpliSeq RNAテクノロジーでは、従来のRNA-Seqと比較して少ないリード数でコストパフォーマンスに優れた網羅的な発現解析を実現することができます。
今回は実際にIon AmpliSeq RNAカスタムパネルを用いて低発現遺伝子のみをピックアップしたパネルを作製し、発現変化量解析を行った事例をご紹介いたします。またIon AmpliSeq Transcriptome Gene Expression KitやqPCRと比較することで、各アプリケーションとの性能の違いについても合わせてお示しします。
<合わせて読みたい:遺伝子パネルがわかる!Ion AmpliSeq テクノロジーのすべて>
<合わせて読みたい:ターゲットシーケンス開発史 Ion AmpliSeq テクノロジー>
AmpliSeq RNAカスタムパネルは、標的遺伝子を絞ることにより低発現の遺伝子でも正確に発現変動を評価可能
今回は以下の条件でIon AmpliSeq RNAカスタムパネル(AmpliSeq RNA)とIon AmpliSeq Transcriptome (ASTx) を用いた発現解析の検証実験を行いました。
■実験条件
サンプル:ヒト肺がん組織・正常組織由来のコントロールTotal RNA(1サンプル10 ng)
パネル:
- Ion AmpliSeq™ Transcriptome Human Gene Expression Kit(A26325)
- Ion AmpliSeq RNAカスタムパネル(AmpliSeq Transcriptomeの解析結果で1~100リードだった48種の遺伝子(内在性コントロールを含む))
ライブラリー調製試薬:
- Invitrogen™ SuperScript™ VILO™ cDNA Synthesis Kit(11754050)(A26325に同梱)
- Ion AmpliSeq™ Library Kit Plus(4488990)(A26325に同梱)
- Ion Xpress™ Barcode Adapters 1-16 Kit(4471250)
(必要試薬の詳細は各ユーザーガイドをご参照ください。)
シーケンス解析にはIon S5™ XL システム (A27214)、Ion Chef™ システム (4484177)を用い、実験手順は各ユーザーガイドに準拠しました。
■解析結果
まず、ASTxとAmpliSeq RNAの解析結果に着目してみましょう。ゲノムにマッピングされた総リード数はAmpliSeq RNAの方が少ないですが、遺伝子を絞っているため全ての遺伝子に対してのリードが取得できています(表2)。一方、ASTxについては3割程度を占める、特に低発現な遺伝子について、十分なリード数が得られないため信頼性の低下する結果となりました。ASTxは幅広い対象遺伝子に対し簡便にスクリーニングするという点で非常に有効な手段ではありますが、AmpliSeq RNAの方が対象遺伝子を絞り込んでいるため、より少ない取得リード数でも目的の遺伝子の詳細な発現解析ができたといえます。また、原理を考慮するとRNA-Seqでは同等のリード数で解析した場合、ASTx以上に発現変動の信頼性の低下した遺伝子が存在する結果が見込まれます。

表2:各パネルを用いた解析結果の概要
10リード以上取得できた遺伝子を検出できた遺伝子と定義
次にASTxとAmpliSeq RNAで遺伝子ごとにリード数がどの程度増えたか比較してみます。
図2はAmpliSeq RNAで解析対象とした48遺伝子の各パネルでの取得リード数を示しています。ASTxで解析した場合の対象48遺伝子の取得リード数は1遺伝子あたり平均45リードでしたが、AmpliSeq RNAでは平均8,544リードと非常に多くのリードが取得できています。

図2:各パネルにおけるAmpliSeqRNAでターゲットとした低発現遺伝子のリード数
遺伝子ごとの実際の値を見てもその差は歴然です。表3は各手法での取得リード数を示しています。例えばLMAN1のAmpliSeq RNAによる取得リード数はASTxの1,000倍程度であることがわかります。サンプルあたりの総リード数はAmpliSeq RNAの方が圧倒的に少ないですが、遺伝子数を48種に絞ったため、ASTxよりも少ない総リード数でも遺伝子ごとに十分なリード数を取得できたといえます。

表3:各パネルの遺伝子ごとのリード数一覧
次に各手法が肺がん組織と正常組織の遺伝子発現量変化を正しくモニターできているか検証を行いました。図3はAmpliSeq RNAでターゲットにした48遺伝子の正常細胞に対する肺がん組織の遺伝子発現量変化を示したグラフです。横軸がAmpliSeq RNA、縦軸がASTxの結果を示しています。ほとんどの遺伝子はどちらの手法でも発現が増加したことを示していますが(図3、赤色の領域)、9遺伝子についてはASTxでは肺がん組織で減少、
AmpliSeq RNAでは増加とそれぞれ異なる結果を示しています(図3、青色の領域)。

図3:肺がん細胞 vs. 正常細胞の遺伝子発現量変化
ASTxおよびAmpliSeq RNAで解析した
では一体どちらの解析結果がより確からしいのでしょうか。これを明らかにするために、該当の9遺伝子を含むApplied Biosystems™ TaqMan™ Arrayプレートを用いてqPCRでも検証しました(図4)。
いずれの遺伝子も肺がん組織で発現量が増加したことを示しており、AmpliSeq RNAの結果と一致していることから、qPCRの結果に基づくとASTxよりもAmpliSeq RNAの方がより確からしい結果であると示唆されます。これはAmpliSeq RNAの方が遺伝子ごとに割り当てられたリード数がASTxより多く、より正確な評価ができたためと考えられます。すなわち、AmpliSeq RNAは、網羅的解析時に問題となりやすい低発現の遺伝子でもより正確に発現量の増減を解析できる手法であるといえます。

図4:AmpliSeq RNAとASTxで発現量変化の増減
一致しなかった9遺伝子のqPCRでの解析結果
Ion AmpliSeq RNAカスタムパネルは誰でも簡単にデザイン可能
以上のようにIon AmpliSeq RNAカスタムパネルは非常に強力な遺伝子発現解析アプリケーションといえますが、オーダー方法も通常のオリゴのように簡便で、アノテーションされた20,000以上の RefSeq遺伝子からターゲットを選択するだけで、誰でも簡単にデザインすることができます。
パネルデザインは以下のようなワークフローで行われます。
- Ion AmpliSeq™ DesignerソフトウエアのWebサイトにサインインします(新規の方はアカウント登録が必要です)。
Ion AmpliSeq Designerソフトウエア:https://ampliseq.com/ - Ion AmpliSeq™ Custom panelsのCreate Custom panelをクリック(図5)。
- Design Nameに任意の名前を入力し、Application TypeはRNA Gene Expression designs (single-pool)、Select genome to useはHuman (RefSeq)を選択(図6)。
※Human以外の生物種を検討されている場合、弊社テクニカルサポート(jptech@thermofisher.com)までご相談ください。 - Next Add Targetsをクリック(図6)。
- 入力フォームに解析したい遺伝子名(Gene Symbol)を入力(遺伝子数が多い場合にはUpload Fileより遺伝子リストをアップロードすると便利です)(図7)。

図5:Ion AmpliSeq Designer ソフトウエア①

図6:Ion AmpliSeq Designer ソフトウエア②
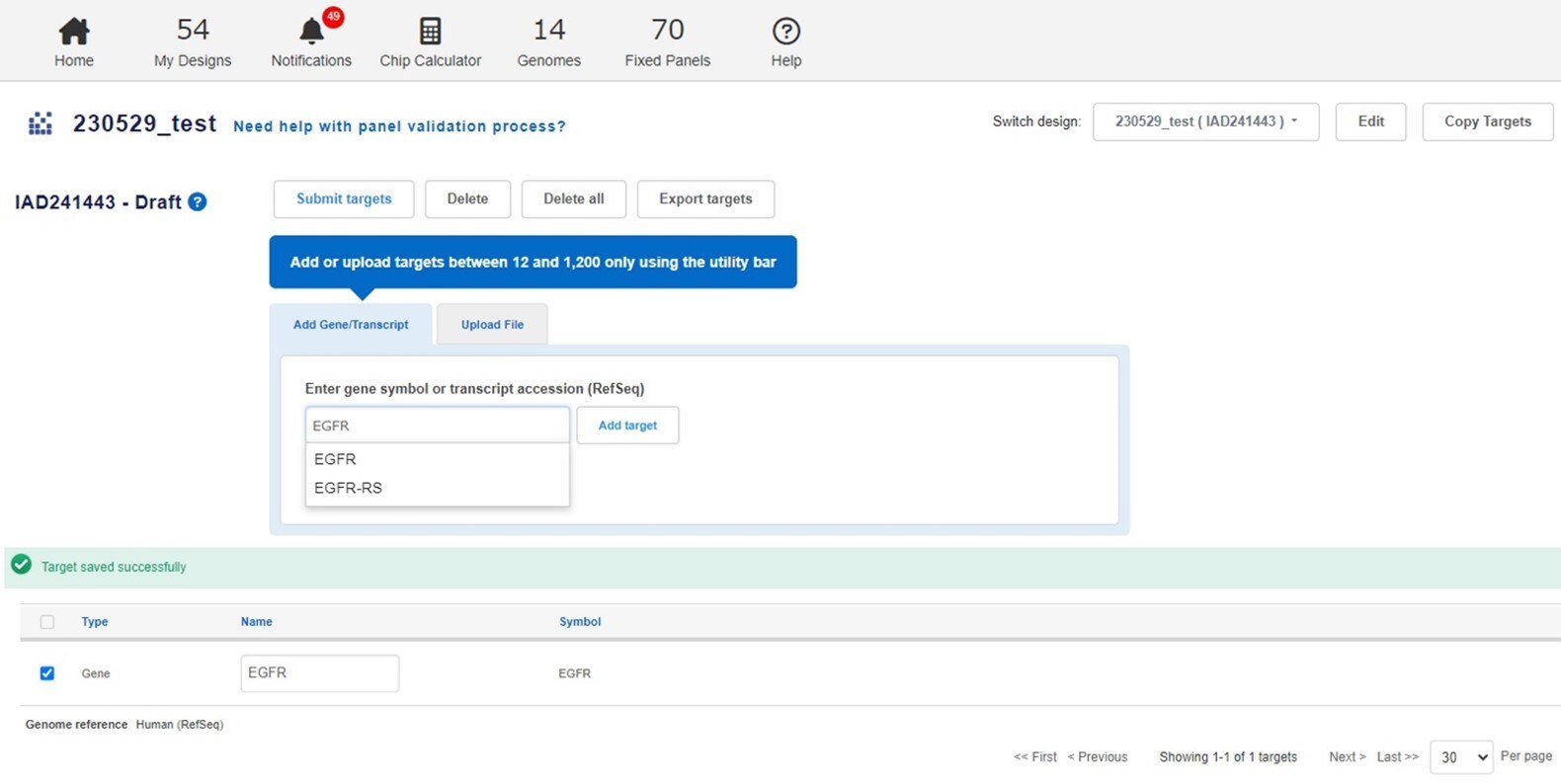
図7:Ion AmpliSeq Designer ソフトウエア③
なお、発注についてはフルアカウント登録が事前に必要になります。
詳細は以下のURLよりご確認ください。
https://www.thermofisher.com/jp/ja/home/technical-resources/order-support/account.html
また完成したパネルは以下の試薬を使用することでライブラリー調製が可能です。
- Ion Torrent™ NGS Reverse Transcription Kit (A45003)など
- Ion AmpliSeq Library Kit Plus (4488990)
- Ion Xpress Barcode Adapters 1-16 Kit (4471250)(複数サンプルを同時に解析する際使用します。)
ライブラリー調製はIon Chef システム (4484177)を用いることで8サンプル同時に自動調製することも可能です。Ion Chefでのライブラリー調製には以下の試薬が必要です。
- NGS Reverse Transcription Kit (A45003)など
- Ion AmpliSeq™ Kit for Chef DL8 (A29024)
詳細については各試薬のユーザーガイドをご確認ください。
Ion AmpliSeq™ Library Kit Plusユーザーガイド
Ion AmpliSeq™ Kit for Chef DL8ユーザーガイド
まとめ
qPCRでは対象となる遺伝子やサンプル数が多すぎる場合、実験に膨大なハンズオンタイムを要します。一方でRNA-Seq法は遺伝子を網羅的に定量できますが、発現変動をくまなく研究するためには膨大なリード数を確保する必要があり、コストと手間がかかります。Ion AmpliSeq RNAテクノロジーでは解析する遺伝子の領域を絞り、さらに対象を絞ることにより1遺伝子に割り当てられるリード数を多く確保でき、低発現遺伝子の場合でもより精度の高い解析が実施可能です。
FFPEサンプルのような微量かつ分解が想定されるサンプルに対してもある程度対応可能ですので、従来のRNA-Seqではライブラリー作製自体が困難であるといった場合にも、お試しいただく価値は十分にあるといえます。
これまでNGSによる発現解析を行ったことがない方や、この記事でIon AmpliSeq RNAテクノロジーに興味を持たれた方が、どのように実験を進めていけばいいか不安に感じていることも多いと思います。弊社ではライブラリー調製方法やIon GeneStudio S5シリーズの操作方法を詳細にお伝えする機会として、ハンズオントレーニングを開催しています。トレーニングの中では実験のポイントを各工程でご紹介していきますので、これから新しくIon Torrent™ 製品を用いたNGS実験を始められる方、あるいは使用経験があってもIon Torrent™ 製品の最新情報や手技を改めて確認したい方など、少しでもご興味がありましたら、まずはお気軽にテクニカルサポートまでご相談ください!
テクニカルサポート連絡先
メール:jptech@thermofisher.com
電話:0120-477-392(ダイレクト番号…NGS実験系3番)
ハンズオントレーニングの一覧
TaqMan is a trademark of Roche Molecular Systems, Inc., used under permission and license.
研究用にのみ使用できます。診断用には使用いただけません。