マイクロアレイを使用した解析が登場してから、既に20年以上が経過しました。この解析技術は現在でも進化を続け、医学分野だけでなく、生命現象に関連した基礎研究分野など、さまざまな目的に応じて使用されています。特に、この解析は遺伝子発現解析に使用されることが多く、1回の実験で特定遺伝子の発現量の増減を測定するNorthern blot法に比べて、数万以上に及ぶ遺伝子の発現量増減を網羅的に測定するスクリーニングツールとしての使用が可能です。
そこで、マイクロアレイによる遺伝子発現解析において、どのような解析が行われてきたかを紹介します。
▼もくじ [非表示]
3’ IVT Arrayによる解析
マイクロアレイ解析が登場した当時の遺伝子発現解析アレイを、ここでは3’ IVT Arrayと総称します。これは、オリゴdTプライマーを利用した逆転写反応とIn Vitro Transcription(IVT)アッセイをベースにしたサンプル調製方法で、下図のようにアレイに搭載される各遺伝子に対応したプローブが、mRNAの3’末端側でデザインされていることに起因しています。

3’ IVT Arrayの使用例
この3’ IVT Arrayを使った解析として、疾患と遺伝子の発現変動の関連性についての研究がありました。その中にはがんを分類したり、あるいはがん患者の予後を予測したりするためのバイオマーカーの検索を行うといったものがあります。例えば、急性リンパ芽球性白血病(ALL)と急性骨髄性白血病(AML)には、それぞれに適切な治療法があるため、分類することが重要です。しかし、この分類には、腫瘍の形態、組織化学、免疫表現型検査、および細胞遺伝学的分析といった、それぞれに専門的な解釈と経験が必要となります。そこでGolubらは、ALLとAMLについてマイクロアレイによる遺伝子発現プロファイルに基づく分子分類の可能性について報告しています1)。初めに既知のALLとAML検体を使って遺伝子発現パターンを確認し、約1,100個の遺伝子発現パターンが相関していることが分かりました。次に別の検体を使って、その確認をすると共にALLとAMLを区別するのに相関性が高い50遺伝子を選択しました。そして、選択した遺伝子の妥当性を検証するために新たな検体で有効性を確認し、この手法を臨床的に実施するためには準備する検体の標準化が必要であることも示唆しています。
また、Wangらは、リンパ節転移陰性の原発性乳がん患者の遠隔転移を予測するバイオマーカーの探索を行っています2)。初めに115人の検体を使って遺伝子発現プロファイルを行い、76個の遺伝子を特定しました。次に別の171人の検体を使って、その確認を行った結果、この遺伝子プロファイルは5年以内に遠隔転移を発症した患者を特定する上で有益であることが分かりました。そしてFoekensらは、複数施設のリンパ節陰性乳がん患者の検体に対して、この76遺伝子プロファイルの検証を行って遠隔転移のある患者を特定するのに有効であることを確認し、補助全身療法を回避できる患者を見分ける可能性があることを示唆しています3)。
一方、このような医学に関係した分野だけでなく、生命現象に関する研究にもマイクロアレイ解析は使用されています。例えば、一般的には体内時計とも言われる概日リズムは多くの生物に存在し、さまざまな生理現象に関わる遺伝子が周期性を持っています。そこで、ゲノム全体が概日リズムによってどのように調整されているのかを把握するためには、マイクロアレイによる網羅的な遺伝子発現解析が適しています。Uedaらは、ショウジョウバエを使って周期的遺伝子発現の網羅的解析を行っています4)。ショウジョウバエ頭部サンプルで解析を行った結果、明暗条件下で712個が周期的に発現変動しており、そのうちの115遺伝子は恒暗条件下でも周期的に発現変動していました。そして、多様な機能に関与する多くの遺伝子が概日リズムの制御下で周期的に発現していて、染色体上でクラスターが存在しており、概日リズム下での遺伝子発現の複雑さを示唆しています。
このように1回の実験で網羅的に遺伝子発現パターンの解析が可能なマイクロアレイ解析は、さまざまな分野で利用されています。そして、これまでの研究で得られた多数のデータは、米国National Center for Biotechnology Information(NCBI)のGene Expression Omnibus(GEO)5)や、英国European Bioinformatics Institute(EBI)のArrayExpress6)のような公共データベースに登録されています。このような公共データベースに登録されているものから該当するデータを利用して一次的な解析を行い、得られた結果について追加の検証実験することで、疾患の予後予測するバイオマーカーを探索したような例もあります7)。
Whole Transcriptome Array(WT Array)による解析
ヒトゲノム解析が進んで遺伝子レベルの研究が発展してくると、ほとんどの遺伝子において選択的スプライシングが起こっていて、さまざまなアイソフォームからなるトランスクリプトが存在することが分かってきました。そのため、例えば遺伝子の3’末端部位を持たないパターンのトランスクリプトが発現していた場合は、これまでの3’末端側にフォーカスしたプローブデザインでのマイクロアレイ解析では検出が困難となります。あるいは、Bcl-x遺伝子のように選択的スプライシングによってアポトーシスを阻害するBcl-xLと促進するBcl-xSが形成され、1つの遺伝子から正負の制御に関係する、相反する因子が作られる場合もあります8)。また、遺伝子の突然変異がスプライシング異常を起こし、そのことが重篤な疾患につながっているとの報告もあります。
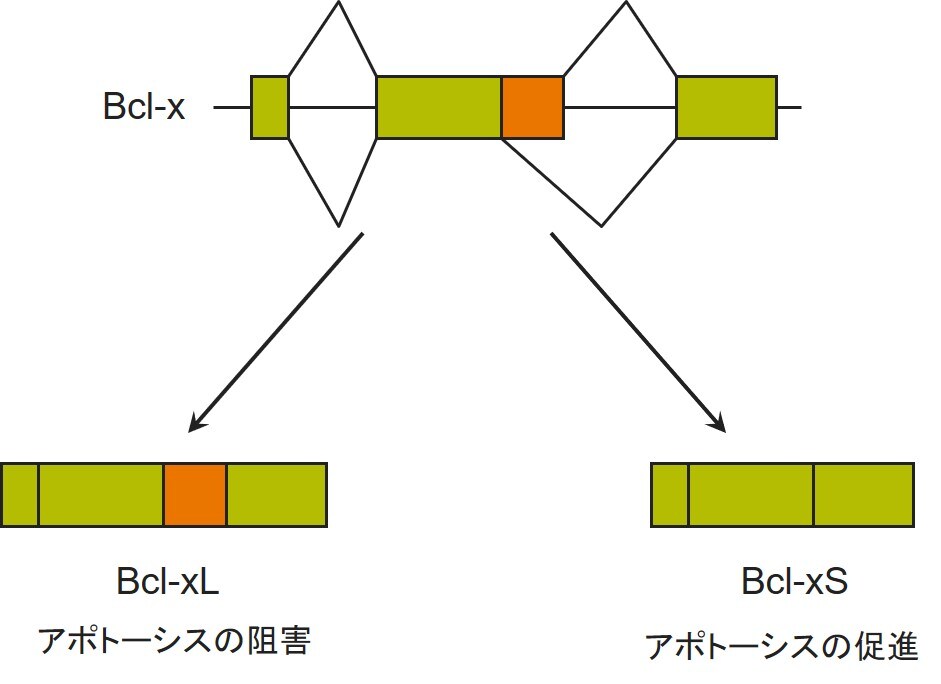

さらに、ゲノム上にはタンパク質をコードしていない長鎖のnon-coding RNA(lncRNA)が多く存在しているということも明らかになり、これらがさまざまな疾患と関連しているという報告もでてきています。
そこで、すべてのトランスクリプトを検出する目的で、プローブデザイン領域を3’末端側だけでなく、トランスクリプト全領域を対象としたWhole Transcript Array(WTアレイ)が登場しました。

研究目的に合わせたClariom S ArrayとClariom D Arrayの特徴と使い分け
現在、ヒト、マウス、ラットの3生物種のWT Arrayは、Applied Biosystems™ Clariom™ S ArrayとClariom™ D Arrayの2種類があります。Clariom S Arrayでは、明確なアノテーション情報を持ったタンパク質をコードしているトランスクリプトをターゲットとした遺伝子レベルの解析を目的としています。一方、Clariom D Arrayでは、タンパク質をコードしているトランスクリプトだけでなく、lncRNAに対するプローブもデザインされているため、どちらのトランスクリプトに対しても発現解析が可能です。さらに、エクソン部分だけでなく、エクソン間のジャンクション部分に対応したプローブもデザインされているため、精度の高いエクソンレベルでの解析も可能です。以上のことから、遺伝子レベルでの発現解析が目的の場合はClariom S Array、遺伝子レベルでの発現解析だけでなくエクソンレベルでの発現解析やlncRNAの発現解析が目的の場合はClariom D Arrayというように、研究目的に合わせてマイクロアレイを利用する形になります。
網羅的な遺伝子発現解析を行う場合、現在は次世代シーケンサーによるRNA-Seqでも対応可能ですが、Clariom S Arrayでは1000万から1億リードのRNA-Seqに相当する解像度のデータを得ることが可能です。また、Clariom D Arrayでは7億リードのRNA-Seqに相当する高解像度のデータを得ることができ、今後のマイクロアレイ解析での主流になることが考えられます。
Clariom S Arrayの使用例
Clariom S Arrayの使用例としては、COVID-19に関連したバイオマーカー探索や治療法への知見についての報告があります。SARS‑CoV‑2に感染した患者間で重症度に違いがあったり、人によっては無症状であったりとさまざまです。Masoodらは、これらの患者からの検体についてApplied Biosystems™ Clariom™ S Array, humanで解析を行い、無症状検体では疾患症候検体と比較すると複数のインターフェロン誘導性遺伝子(ISG)が有意に発現増加していることが分かりました9)。そのため、無症状の場合のSARS‑CoV‑2感染後初期では、ISGによって効果的に制御されている可能性が示唆されました。そのことから、COVID-19の初期段階ではISGを活性化するI形インターフェロンによる治療は、疾患の進行を抑制する可能性があると考えられました。
Clariom D Arrayの使用例
一方、Clariom D Arrayの使用例としては、腫瘍細胞では異常なスプライシングにより、本来のトランスクリプトとは異なったアイソフォームが発現していることがあります。Taniguchi-Poncianoらは、Applied Biosystems™ Clariom™ D Array, humanを使って2種類の細胞系統(POU1F1、NR5A1)に由来する下垂体腺腫のトランスクリプトーム解析を行いました10)。その結果、POU1F1細胞系統由来の下垂体腫瘍ではTHY1、BMPER、GRIA3などの遺伝子で選択的スプライシングによるアイソフォームが生成していました。一方、NR5A1細胞系統由来の下垂体腫瘍ではCDK18、SEMA7A、PTGS2、KCNA3などの遺伝子の選択的スプライシングによるアイソフォームが生成していました。また、下垂体腫瘍をスプライシング関連遺伝子プロファイルによって分類できることも推察しています。これらのことから、アイソフォームやスプライシング関連遺伝子のプロファイルは、腫瘍のサブタイプを特徴づけ、分子マーカーおよび分子療法の標的として使用できる可能性があると示唆しています。
また、lncRNAをターゲットにした研究にもClariom D Arrayは使用されています。例えば、加齢に伴って起こる嗅覚機能の低下は高齢者の生活の質に影響を及ぼし、潜在的な神経変性疾患の早期臨床症状の1つとして考えられています。そこで、Wangらは、年齢の異なる3群のマウスをモデルにしてApplied Biosystems™ Clariom™ D Array, mouseを使い、嗅覚障害の潜在的役割に関わる因子を検索しました11)。その結果、若い成体マウスと老齢マウスでは嗅覚情報伝達で中心的な役割を持っている嗅球において、112個のタンパク質コーディング遺伝子(PCG)と84個のlncRNAが発現変動していました。これらのPCGは、シグナル伝達、遺伝子発現調節、細胞微小環境に関連していました。また、2つのlncRNA(NONMMUT004524、NONMMUT000384)が老齢マウスの嗅球における神経活性リガンド-受容体相互作用経路と有意に関連しており、潜在的な悪影響を与えている可能性があることを示唆しています。
miRNA Arrayによる解析
miRNAは20~25塩基程度の長さからなる一本鎖RNAで、さまざまな生物に存在することが知られているタンパク質には翻訳されないnon-coding RNAの1つです。この鎖長の短いRNA分子は、さまざまなmRNAの発現を調節し、分化、細胞増殖、アポトーシスなど、生物にとって欠かせない生命現象に関わっていると考えられています12), 13)。また、がんなどの疾患においてmiRNAの発現プロファイルが固有のパターンを示していることが分かり、miRNAをバイオマーカーとして検討する研究が増えてきています14), 15)。
GeneChip miRNA Arrayの使用例
Applied Biosystems™ GeneChip™ miRNA Arrayの使用例として、Yongらは、組織と血液の2つの検体を使って結腸直腸がん(CRC)の早期発見のためのバイオマーカー探索を行っています16)。その結果、両タイプの検体に共通して7種類のmiRNA(miR-150、miR-193a-3p、miR-23a、miR-23b、miR-338-5p、miR-342-3p、miR-483-3p)に発現変動が見られ、さらにリアルタイムPCRでの検証で、3種類のmiRNA(miR-193a-3p、miR-23a、miR-338-5p)は、組織および血中レベルで有意に正の相関があることが観察されました。これらのmiRNAの発現増加は、がんがより進行した段階でも検出されており、CRC早期発見のためにリキッドバイオプシーとしての血液検体からのバイオマーカーとなる可能性を示唆しています。
また、Kumarらは、アルツハイマー病患者と軽度認知障害患者と健常人からの血清検体を用いてマイクロアレイ解析を行い、その結果を元にリアルタイムPCRでの検証を行った結果、miR-455-3pとmiR-4668-5pの2種類のmiRNAが有意に発現増加していることが分かりました17)。さらにトランスジェニックマウスにおいても、アルツハイマー病の影響を受けている脳の大脳皮質領域では、mmu-miR-455-3pが高レベルであるのに対し、影響を受けていない小脳領域では低レベルであることが明らかになりました。また、パスウェイ解析からmiR-455-3pはいくつかの生物学的経路の調節に関連していて、これらの経路に関連する遺伝子はアルツハイマー病の病因に重要な役割を果たしていることが分かりました。 以上のことから、アルツハイマー病患者に見られるmiR-455-3pの発現増加は、バイオマーカーとしての役割を示していることを示唆しています。
まとめ
以上、遺伝子発現解析におけるマイクロアレイの活用事例について紹介してきました。
GeneChip 3’ IVT Arrayは、マイクロアレイ解析が登場してからのデザインを踏襲しており、論文数やデータベースへの登録数も数多いため、それらのデータベースを利用したり、過去の例とご自身の研究を比較したりする場合に適しています。また、Clariom ArrayのようなWT Arrayは、全転写領域をターゲットにした新しいデザインからなり、新規に研究を始める場合におすすめです。一方、GeneChip miRNA Arrayは、多くの生命現象に関わる鎖長の短いnon-coding RNAをターゲットにした研究に利用できます。それぞれの製品については以下のリンクで確認できます。
・GeneChip™ Human Genome U133 Plus 2.0 Array
・Clariomアッセイ
・マイクロアレイを用いた miRNA プロファイリング
この事例で使用しているマイクロアレイは、弊社が提供しているフリーの解析ソフトウエア、Applied Biosystems™ Transcriptome Analysis Console(TAC)ソフトウエアを使えば、バイオインフォマティクスに精通していなくても簡単にデータ解析を行うことができます。
また、マイクロアレイによる解析は、今回紹介しました遺伝子発現解析のようなRNA解析だけでなく、染色体DNAを使ったSNP genotypingからのGenome-Wide Association Study(GWAS)による疾患原因遺伝子領域の探索や、染色体のコピー数異常を検出する染色体コピー数解析にも利用可能で、さまざまな研究に対応しています。こちらについては、改めて紹介できたらと考えています。
参考文献
1)Golub, T.R. et al. Science 286, 531-537, 1999
2)Wang, Y. et al. Lancet 365, 671-679, 2005
3)Foekens, J. A. et al. J. Clin. Oncol. 24, 1665-1671, 2006
4)Ueda, H. R. et al. J. Biol.Chem. 277, 14048-14052, 2002
5)Gene Expression Omnibus, https://www.ncbi.nlm.nih.gov/geo/
6)ArrayExpress, https://www.ebi.ac.uk/arrayexpress/
7)Naoi, Y. et al. Breast Cancer Res. Treat. 128, 633-641, 2011
8)Lawrence, H. B. et al. Cell 74, 597-606, 1993
9)Masood, K. I. et al. Scientific Reports 11, 22958, 2021
10)Taniguchi-Ponciano, K. et al. Genes 11, 1422, 2020
11)Wang, M. et al. Scientific Reports 7, 2079, 2017
12)Ambros, V. Nature 431, 350-355, 2004
13)Murphy, D. et al. BMC Evol. Biol. 8, 92, 2008
14)Mitchell, P. S. et al. Proc. Natl. Acad. Sci. USA 105, 10513–10518, 2008
15)Montani, F. et al. J Natl Cancer Inst. 107, djv063, 2015
16)Yong, F. L. et al. BMC Cancer 13, 280, 2013
17)Kumar, S. et al. Human Mol. Genet. 26, 3808-3822, 2017
研究用にのみ使用できます。診断用には使用いただけません。



